УЧРЕЖДЕНИЕ РОССИЙСКОЙ АКАДЕМИИ НАУК
САНКТ-ПЕТЕРБУРГСКИЙ АКАДЕМИЧЕСКИЙ УНИВЕРСИТЕТ—
НАУЧНО-ОБРАЗОВАТЕЛЬНЫЙ ЦЕНТР НАНОТЕХНОЛОГИЙ РАН
На правах рукописи
Диссертация допущена к защите
Зав. кафедрой
А.В. Омельченко
"" 2013 г.
ДИССЕРТАЦИЯ
НА СОИСКАНИЕ УЧЕНОЙ СТЕПЕНИ
МАГИСТРА
Тема: Использование неравномерного покрытия ридами генома для разрешения повторов при секвенировании генома одной клетки Направление: 010900.68 — Прикладные математика и физика Магистерская программа: "Математические и информационные технологии" Выполнила студентка К.В.Крашенинникова Руководитель Д.Ю.Антипов Рецензент Н.И.Вяххи Санкт-Петербург Реферат C. 22, рис. 6, табл. 1.Настоящая работа содержит описание подхода к разрешению повторов при сборке геномов на основе данных секвенирования генома из одной клетки. Приведено описание различных этапов работы алгоритма. Описанный метод реализован и интегрирован в геномный ассемблер SPAdes. Рассмотрены также другие существующие методы разрешения повторов при сборке геномов. Проведен сравнительный анализ работы других методов и предлагаемого подхода и приведены результаты сравнения.
Ключевые слова: сборка геномов, разрешение повторов.
Содержание Терминологический словарь Введение Сборка генома Подходы к разрешению повторов Алгоритм разрешения повторов Реализация описанного подхода Результаты Заключение Список литературы Терминологический словарь Геном — cовокупность наследственной информации организма, обычно подразумевается совокупность последовательностей ДНК.
В рамках настоящей работы под геномом мы будем подразумевать строку над четырехбуквенном алфавитом нуклеотидов {,,, }.
Контиг — выдаваемая ассемблером последовательность нуклеотидов, являющихся подстроками генома.
Нуклеотид — молекула, состоящая из азотистого основания, сахара и фосфатной группы. Следующие нуклеотиды входят в состав ДНК: аденин (А), цитозин (С), гуанин (G), тимин (Т).
В рамках настоящей работы под нуклеотидами мы будем понимать буквы алфавита {,,, }.
Рид — подстрока генома, полученная в результате секвенирования.
Покрытие участка генома ридами — количество ридов, полученных в результате секвенирования данного участка генома.
Праймер — короткий фрагмент нуклеиновой кислоты, комплементарный некоторому участку шаблонной ДНК. Используется как последовательность, с которой начинается синтез комплементарной цепочки ДНК.
Секвенирование — биологический процесс определения последовательности нуклеотидов. Результатом секвенирования являются риды.
Введение Секвенирование и сборка геномов Дезоксирибонуклеиновая кислота (ДНК) была открыта в году Иоганном Фридрихом Мишером. В 40-х - 50-х годах XX века было установлено, что молекула ДНК является носителем наследственной информации. А в 1953 году Джеймс Уотсон и Френсис Крик предположили, что ДНК имеет форму двойной спирали [8]. Эти открытия предопределили дальнейшие направления исследований по изучению механизмов хранения и передачи наследственной информации. В настоящее время этой областью исследований решаются следующие задачи:
диагностика генетических заболеваний производство лекарственных средств определение эволюционного родства между организмами генная инженерия Современные технологии позволяют определять первичную структуру геномов (читать геномы) длиной в миллиарды нуклеотидов.
Первые исследования в этой области начались в 70-х годах XX века.
В 1976 году группой ученых во главе с Уолтером Фирсом был определен первый полный геном вируса - бактериофага MS2.
Одной из ранних технологий, позволяющих прочесть геном, была технология, предложенная Фредериком Сэнгером в 1977 году. Подход Сэнгера заключается в следующем: одноцепочечные молекулы ДНК помещаются в раствор с полимеразой и обычными деоксинуклеотидами (аденин, цитозин, тимин и гуанин). Затем поэтапно в раствор добавляются дидеоксинуклеотиды. Полимеризация этих молекул с ДНК завершает процесс элонгации, то есть построения двухцепочечной молекулы ДНК. После нескольких серий добавления дидеоксинуклеотидов, полученные молекулы разделяются по длине с помощью гель-электрофореза, и таким образом определяется, в какой последовательности дидеоксинуклеотиды полимеризовались с ДНК.
Эта технология обладает высокой точностью (99.9%); она доминировала среди методов секвенирования на протяжении 25 лет. Однако, несмотря на то, что технология Sanger используется и в наши дни, она весьма дорогостоящая.
В 1990 году был запущен проект Human Genome, целью которого было определить геном человека. В 2003 году этот проект был завершен, в результате получена последовательность человеческого генома, состоящая из трех миллиардов нуклеотидов. Впоследствии были получены новые сборки генома человека, которые содержат исправления и уточнения последовательности. Проведенное исследование послужило толчком для развития персональной медицины, основанной на использовании генетической информации каждого индивидума в отдельности. Однако высокая стоимость секвенирования служила серьезным препятствием на пути развития и популяризации этого направления. В результате были разработаны новые технологии, с помощью которых можно было бы читать большие геномы, состоящие из миллиардов нуклеотидов. На протяжении последних нескольких лет одной из самых популярных технологий секвенирования является технология Illumina, которая позволяет получать достаточно точные данные (98%), при этом стоимость чтения одного нуклеотида в десятки раз меньше, чем стоимость данных, полученных по методу Сэнгера.
В процессе чтения генома полученные данные представляют собой множество фрагментов геномной последовательности. В результате возникает задача ассемблирования, то есть сборки геномной последовательности из полученных фрагментов.
Кроме технологий Illumina и Sanger, существуют также другие подходы к секвенированию, например, 454, PacBio, Ion Torrent. Все методы секвенирования позволяют получать фрагменты генома разной длины и характеризуются различной точностью данных. Этапом, предшествующем любому современному методу секвенирования, является предварительное накопление биологического материала в количестве, достаточном для секвенирования. Самый распространенный метод накопления генетического материала - это культивация клеток, то есть их выращивание в специальных условиях.
Однако существующие технологии не применимы к некоторым видам клеток. В настоящее время невозможно культивировать большинство бактерий, а также мутирующие клетки раковых опухолей многоклеточных организмов. Для решения этой проблемы были разработаны технологии амплификации ДНК, полученной из единственной клетки.
Наиболее распростаненной технологией в области секвенирования генома одной клетки является multiple displacement amplification (MDA).
Эта технология была предложена группой ученых во главе с Роджером Ласкеном [3] в 2001 году. Одной из особенностей данных, полученных по технологии MDA, является неравномерность покрытия генома прочитанными фрагментами. Как правило, это свойство приводит к затруднениям при сборке, так как традиционно сборщики пользуются информацией о покрытии для фильтрации ошибочных данных. Тем не менее, существуют ассемблеры (SPAdes [1], IDBA-UD [5], Velvet-SC [2]), то есть геномные сборщики, которые учитывают особые свойства данных, полученных по этой технологии.
После того, как получено достаточно копий генетического материала, производится секвенирование. В настоящей работе рассматривается подход к сборке данных, полученных в результате применения технологий MDA и Illumina.
Фрагменты ДНК, полученные по технологии Illumina, имеют в среднем длину порядка 100 - 250 нуклеотидов. Фрагменты, полученные по этой технологии, короче фрагментов, полученных по технологии Sanger (400-900 нуклеотидов), что значительно усложняет задачу сборки. Распростаненным подходом для сборки таких коротких последовательностей является метод, основанный на постоении графа Де Брюйна. Этот подход был предложен Павлом Певзнером в году. Для этого из полученных фрагментов генома выделяются подпоследовательности фиксированной длины. На основе информации о перекрытиях между выделенными подпоследовательностями строится граф. В результате задача о сборке генома сводится к задаче нахождения покрывающих путей в графе.
Разнообразие структуры геномов приводит к различным сложностям при сборке. В большинстве геномов одноклеточных и многоклеточных живых организмов существуют повторяющиеся элементы. Это явление приводит к появлению нескольких возможных вариантов путей в графе, выбор правильного из них называется разрешением повтора. Однако, для того, чтобы выбрать верный путь в графе, нужна дополнительная информация. Как правило, используются данные, содержащие парные риды, то есть риды, расстояние между которыми известно с некоторой точностью. Если некоторой паре ребер в графе можно сопоставить пару фрагментов, расстояние между которыми известно, то с помощью этой информации можно определить правильный маршурт в графе и разрешить повтор. Однако не все существующие секвенаторы позволяют получать данные с парной информацией. Подход, описанный в нашей работе, позволяет разрешать повторы без использования дополнительных данных.
Вместо этого мы предлагаем анализировать информацию о количестве геномных фрагментов, соответствующих определенным ребрам полученного графа.
Секвенирование генома одной клетки. Технология MDA Существует несколько технологий секвенирования генома одной клетки, например, MDA [3] и MALBAC. Технология Multiple Displacement Amplification (MDA) является де-факто стандартом выделения ДНК в этой области.
Метод MDA основан на применении полимеразы Phi 29. Эта полимераза позволяет строить комплементарные цепочки длиной 7- тысяч нуклеотидов. Процесс начинается с лигирования (присоединения) праймеров к одной из цепочек ДНК. Начиная с праймеров, полимераза достраивает комплементарную цепь ДНК. Когда синтез ДНК достигает следующего праймера, процесс гибридизации завершается и полимераза отсоединяет вновь построенную цепочку ДНК от исходной. Таким образом удается увеличить количество одноцепочечных ДНК и провести последовательно несколько этапов гибридизации и в результате получить большее количество генетического материала.
Секвенирование данных, полученных по технологии MDA, влечет за собой особые свойства получаемых данных:
Неравномерность покрытия генома ридами Большое количество ошибочных (химерных ) ридов, содержащих фрагменты непоследовательных участков генома Как правило, покрытие генома данными, полученными в результате традиционного секвенирования, достаточно равномерно. Поэтому информацию о покрытии используют для того, чтобы отсеять ошибочные данные. В результате MDA разные участки генома амплифицированы разное количество раз, что является причной неравномерного покрытия генома. Таким образом, затруднительно использовать информацию о покрытии для фильтрации ошибочных данных.
Однако, можно предположить, что участки генома, расположенные близко друг к другу, имеют более близкое покрытие, чем те, которые расположен далеко друг от друга. В настоящей работе неравномерность покрытия генома используется для разрешения повторов.
Сборка генома Задача сборки геномов Данные секвенирования можно рассматривать как множество строк из алфавита {,,, }, которые являются подстроками строки, соответствующей геному. В результате возникает задача сборки генома, то есть поиска строки, которая содержала бы риды и как можно точнее соответствовала бы исходному геному. Различают два вида сборки: de novo сборка и сборка на основе вспомогательного (референсного) генома. Во втором случае в качестве референса используют уже собранный геном такого же организма или эволюционно близкого другого организма. Далее в настоящей работе будет идти речь только о подходе de novo.
В качестве математической модели задачи принято рассматривать задачу о поиске кратчайшей общей надстроки (SSP, Shortest Superstring Problem), которая формулируется следующим образом.
Дано множество строк = {1, 2,..., } над алфавитом. Необходимо найти строку минимальной длины, такую что любая строка из является подстрокой. Доказано, что в такой постановке задача о сборке является -полной [4].
Таким образом, если =, не существует точного полиномиального алгоритма, который решал бы поставленную задачу.
Описанная модель основана на двух предположениях. Первое предположение заключается в том, что каждый рид должен являться подстрокой генома. Однако в датасетах встречаются ошибочные риды и контаминации, содержащие генетическую информацию сторонних организмов.
Кроме того, в геномах большинства организмов существуют повторы - несколько идентичных или почти идентичных участков генома.
Подход, реализующий концепцию SSP, будет сопоставлять всем таким повторам одну единственную последовательность в геноме. В результате задача сборки генома усложняется.
Граф де Брюйна NP-трудность задачи о надстроке не исключает того, что у данной задачи есть частные случаи, для которых существуют эффективные алгоритмы. Предположим, что длины всех строк одинаковы и зафиксируем длину возможного пересечения строк. В качестве математической модели, совместимой с такими ограничениями, выберем граф де Брюйна [6], [9]. Рассмотрим множество = {1, 2,..., } всех ридов. Зафиксируем некоторое число k, которое не превосходит минимальной из длин ридов. Каждый рид можно представить как множество строк длины k над алфавитом {,,, }. Такие строки будем называть k -мерами. Построим граф над множеством всех k -меров следующим образом. Каждому k -меру сопоставим вершину. Любую пару k -меров и, имеющих общую подстроку длины k-1, таких что последние k-1 нуклеотидов совпадают с первыми k- нуклеотидами, соединим направленным ребром (, ).
Любой путь в графе соответствует некоторой строке. В том числе, если предположить, что риды не содержат ошибок и покрывают геном полностью, исходный геном будет соответствовать некоторому пути в графе.
Часто построенный таким образом граф де Брюйна можно упростить, объединяя ребра, принадлежащие одной цепи. То есть если входящая и исходящая степени некоторой вершины равны 1, то соответствующее входящее и исходящее ребра объединяются в новое ребро. Граф, получившийся в результате таких преобразований, называется сжатым. При объединении ребер соответствующие им последовательности объединяются. В результате последовательности ребер в сжатом графе могут иметь любую длину, не меньше k+1.
От выбора значения k зависит чувствительность нашей модели к коротким пересечениям ридов. Так, если выбрать k слишком большим, то это может привести к игнорированию пересечений длины короче, чем k-1. С другой стороны, если k выбрать слишком маленьким, то это может привести к усложнению структуры графа и неспособности без дополнительной информации однозначно разрешать маршруты через повторные последовательности, встречающиеся в геномах. Некоторые ассемблеры (SPAdes, IDBA, IDBA-UD) для разрешения этой проблемы варьируют k, но этого все равно недостаточно, чтобы разрешить все неоднозначности такого рода. В результате это приводит к фрагментации последовательности. То есть вместо одной строки, соответствующей геному, возможно собрать только некоторое количество строк, называемых контигами, соответствующих разным путям в графе и разным фрагментам генома. Настоящая работа посвящена одному из подходов, позволяющих разрешать повторы, вызывающие неоднозначности при обходе графа, и таким образом, увеличивать длину строк, соответствующих непрерывным участкам генома.
Геномные повторы в графе де Брюйна Большинство изученных геномов различных организмов содержат повторы. Среди них выделяют особый вид повторов, которые встречаются в геноме несколько раз подряд. Такие повторы называют тандемными. В зависимости от длины повторяющейся последовательности, тандемные повторы называются сателлитными (> нуклеотидов), минисателлитными (10-60 нуклеотидов) и микросателлитными (7%), поэтому сборка генома из таких данных требует особого подхода.
Однако информации, содержащейся в таких ридах, достаточно для того, чтобы приложить их к ребрам сжатого графа де Брюйна, построенного на основе коротких ридов. В том случае, если длинный рид прикладывается к паре ребер, одно из которых входит в повтор, а второе выходит из повтора, можно определить маршрут в графе, соответствующий правильной геномной последовательности. Таким образом, можно разрешать повторы, длины которых не превышают длину последовательности длинных ридов.
Использование информации о покрытии ридами генома Информация о покрытии ридами генома может использоваться в случае ее неравномерного распределения для разных позиций генома, как в случае данных, полученных в результате секвенирования генома по технологии MDA.
Подход, описанный в настоящей работе, основан на следующем предположении: несмотря на то, что распределение покрытия по геному в целом неравномерно, участки, расположенные в геноме близко друг другу, будут иметь схожее покрытие.
Понятие покрытия может быть также определено для ребер сжатого графа де Брюйна. Под кратностью k -мера мы понимаем количество вхождений k -мера в риды. Тогда значением покрытия ребра будет называться среднее значение кратности k -меров, образующих последовательность ребра.
Однако ребра в сжатом графе могут иметь длину порядка десятка тысяч нуклеотидов. Покрытие генома на данных MDA может сильно меняться на таком промежутке. По этой причине в настоящей работе мы будем пользоваться также понятием входящего и исходящего покрытия.
Рассмотрим некоторое ребро сжатого графа де Брюйна длины.
Зафиксируем два целых числа, <. Входящим покрытием ребра будем считать среднюю кратность первых k -меров, образующих последовательность ребра. Аналогично исходящим покрытием ребра будем считать среднюю кратность последних k -меров, образующих последовательность ребра.
Алгоритм разрешения повторов Выделение повторных компонент В качестве входящей информации мы используем граф де Брюйна, информацию о входящем и исходящем покрытии для каждого ребра, а также парную информацию. Нашей целью является разрешить потвторы и в результате получить пути, проходящие четрез повторы, которые содержат входящее ребро, повтор и соответствующее исходящее ребро.
Наш подход состоит из двух этапов.
Первый этап заключаются в определении ребер, которые соответствуют геномным повторам. На втором этапе происходит сопоставление ребер, входящих в повтор, исходящим ребрам. Под повторной компонентой мы будем понимать группу смежных ребер, соответствующих повторяющимся участкам генома. Разделим все ребра сжатого графа на одиночные, то есть соответствующие уникальным геномным последовательностям, и повторные.
Выделение повторных компонент с помощью анализа топологии графа Для каждого ребра = (, ) в графе рассмотрим его исходящую и входящую вершины. Ребро будем считать одиночным, если выполняются оба условия:
В том случае, если обрабатывать приходится сжатый граф, не содержащий ребер, соответствующих ошибкам секвенирования, такой подход определения повторов работает в большинстве случаев.
Тем не менее, можно привести примеры, когда описанный подход не Рис. 4: An image of a tip (red vertex is a terminal one) and a bulge Рис. 5: Определение повторов с помощью парной информации. Исходя из топологии, ребро = (7, 8) в графе невозможно определить как повторное, но парная информация позволяет классифицировать его правильно.
позволит правильно детектировать все повторные ребра (рис.5).
Выделение повторных компонент с помощью парной иформации Рассмотрим две пары парных ридов (1, 2 ) и (1, 2 ). Пусть риды 1 и 1 прикладываются к соответствующим последовательностям некоторого ребра в сжатом графе де Брюйна (рис.5), а риды 2 и 2 прикладываются к последовательностям некоторых ребер 1 и соответственно. Тогда ребро будем считать повторным в том случае, если не существует достаточно короткого пути в графе между ребрами 1 и 2.
Разрешение повторов После того, как все ребра в графе разделены на одиночные и повторные, можно приступать к разрешению повторов. Для этого у каждой повторной компоненты выделяется группа входящих и группа исходящих ребер. В том случае, если количество элементов в обеих группах различно, повтор не будет разрешен, потому что такая ситуация свидетельствует о том, что повтор определен неправильно. Кроме того, такой случай возможен, если в графе существуют ошибочные (химерные) ребра (не соответствующие настоящим геномным последовательностям собираемого организма).
Для того, чтобы разрешить повтор, каждая группа из входящих и исходящих ребер {1, 2,..., } и {1, 2,..., } сортируется в порядке убывания значений покрытия (при более детальном подходе группа входящих ребер сортируется по убыванию значений входящего покрытия, а группа исходящих ребер - исходящего).
Возможны ситуации, когда мы не можем быть уверены, что повтор будет разрешен правильно. Такие случаи могут быть вызваны близкими значениями покрытия между элементами одной и той же группы или слишком разными значениями покрытия у пары сопоставляемых ребер. Для того, чтобы избегать таких ситуаций, мы вводим отсечки, которые ограничивают значения отношений между соответствующими значениями покрытий:
Рис. 6: Пример структуры на сжатом графе де Брюйна, соответствующей сложному тандемному повтору.
Обработка тандемных повторов Тандемный повтор представляет собой последовательность нескольких повторяющихся подряд одинаковых строк, поэтому под разрешением повтора в данном случае понимается определение количества таких повторений в пределах одного тандемного повтора.
Тандемные повторы представляют собой достаточно короткие последовательности, поэтому в данном случае можно предположить, что покрытие участка генома, в котором помещается тандемный повтор, будет достаточно равномерным. Мы воспользуемся этим, чтобы разрешать тандемные повторы по кратности покрытия.
Ребра, входящие в повторы, образуют на графе компоненты сильной связности. Мы определяем их с помощью двунаправленного поиска в глубину. Полученное в результате множество компонент мы разделим на две группы, соответствующих простым и сложным (рис.6) тандемным повторам. Тандемный повтор будем называть сложным, если он содержит в себе другой тандемный повтор.
Простые тандемные повторы образуют на графе циклы из пары ребер. Их можно разрешить по кратности покрытия.
Сложные повторы образуют более сложные циклы на графе и не всегда однозначно разрешаются по кратности покрытия, поэтому мы просто не рассматриваем такие ребра для разрешения повторов, описанного в предыдущих пунктах.
Реализация описанного подхода Геномный ассемблер SPAdes Ассмеблер SPAdes [1] предназначен для сборки бактериальных геномов на основе данных одноклеточного секвенирования. Рабочий цикл SPAdes состоит из нескольких этапов:
1. Коррекция ошибок в ридах 2. Построение сжатого графа 3. Упрощение структуры сжатого графа (удаление пузырей, тупиков и химерных ребер) 4. Разрешение повторов на основе парной информации Наш подход разработан как отдельный модуль, который интегрируется в рабочий цикл ассемблера SPAdes перед этапом разрешения повторов по парной информации, то есть между этапами 3 и 4 рабочего цикла. Кроме того, наш модуль может использоваться самостоятельно, независимо от других модулей, разрешающих повторы на графе.
Результаты Чтобы оценить эффективность описанного подхода к разрешению повторов, было проведено сравнение результатов сборки ассемблера SPAdes в сочетании с модулем, реализующим наш метод, и без него. Результаты оценки характеристик различных сборок были получены с помощью программы QUAST и приведены в табл. Были учтены следующие характеристики:
# contigs - количество контигов длины 500 нуклеотидов;
NGA50 - такая длина фрагмента, что более 50% референсного генома покрыто фрагментами контигов, равной или большей Largest contig - длина самого длинного контига;
Genome mapped - отношение покрытых сборкой нуклеотидов к длине референсного генома;
MA - количество мизассемблов, то есть неправильных соеднинений геномных последовательностей;
# genes - общее количество генов, полностью присутствующих Видно, что те запуски, в которых использовалось разрешение повторов по покрытию, демонстрирует лучшие результаты.
Таблица 1: Сравнение сборок датасета E. coli single-cell. Жирным шрифтом выделены запуски, включающие модуль разрешения повторов по покрытию (single reads) + cov-based-rr (single reads) +Path-Extend (paired reads) + Path-Extend + cov-based-rr (paired reads) Заключение Результаты работы Результатом работы является программный модуль, интегрированный в геномный ассмеблер SPAdes. Была продемонстирована эффективность выбранного подхода для разрешения повторов при сборке данных, полученных при секвенировании генома одной клетки.
Исследование было представлено в рамках стендового доклада на Международной конференции по вычислительной биологии RECOMB 2013.
Список литературы [1] Anton Bankevich, Sergey Nurk, et al. SPAdes: A New Genome Assembly Algorithm and Its Applications to Single-Cell Sequencing.
Journal of computational biology : a journal of computational molecular cell biology, 19(5):455–477, May 2012.
[2] et. al Chitsaz. Efficient de novo assembly of single-cell bacterial genomes from short-read data sets. page 915–921, 2011.
[3] Roger S. Lasken. Single-cell genomic sequencing using Multiple Displacement Amplification. Current opinion in microbiology, 10(5):510–516, October 2007.
[4] Paul Medvedev, Konstantinos Georgiou, Gene Myers, and Michael Brudno. Computability of models for sequence assembly. pages 289– 301, 2007.
[5] Yu Peng, Henry C. M. Leung, Siu-Ming Yiu, et al. IDBA - A Practical Iterative de Bruijn Graph De Novo Assembler. In Research in Computational Molecular Biology, 14th Annual International Conference, RECOMB 2010, Lisbon, Portugal, April 25-28, 2010.
Proceedings, volume 6044 of LNCS, pages 426–440, 2010.
[6] Pavel A. Pevzner, Haixu Tang, and Michael S. Waterman. An Eulerian path approach to DNA fragment assembly. Proceedings of the National Academy of Sciences, 98(17):9748–9753, August 2001.
[7] Ussery et al. Genome update: Dna repeats in bacterial genomes.
Microbiology, 150(11 3519-3521), November 2004.
[8] J. D. Watson and F. H. C. Crick. A structure for deoxyribose nucleic acid. pages 737–738, 1953.
[9] Daniel R. Zerbino and Ewan Birney. Velvet: Algorithms for de novo short read assembly using de Bruijn graphs. Genome Research, 18(5):821–829, May 2008.

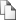
 [
[  «
«