На правах рукописи
Жмуров Артём Андреевич
Моделирование больших биомолекул и
биомолекулярных систем
с использованием графического
процессора
Специальность 05.13.18 – математическое
моделирование, численные методы и комплексы
программ
Автореферат диссертации на соискание учёной степени
кандидата физико-математических наук
Москва - 2011
Работа выполнена на кафедре вычислительной математики Московского физико-технического института (государственного университета)
Научный руководитель:
кандидат физико-математических наук, доцент Холодов Ярослав Александрович
Официальные оппоненты:
доктор физико-математически наук, профессор Ефремов Роман Гербертович доктор физико-математически наук, профессор, кандидат биологических наук Пантелеев Михаил Александрович
Ведущая организация:
Московкий государственный университет им. М.В. Ломоносова, Кафедра биофизики
Защита диссертации состоится “27” октября 2011 г. в 1200 час. на заседании диссертационного совета Д 212.156.05 при Московском физико-техническом институте (государственном университете) по адресу: 141700, г. Долгопрудный Московской обл., Институтский пер. д.9, ауд. 903 КПМ.
С диссертацией можно ознакомится в библиотеке МФТИ (ГУ).
Автореферат разослан “26” сентября 2011 г.
Учёный секретарь диссертационного совета Д 212.156.05 Федько О.С.
Общая характеристика работы
Актуальность темы. Большие белковые молекулы, образования и волокна играют важную роль в жизнедеятельности организма. Нити фибрина, образуя сложную ветвистую структуру, формируют сгусток и останавливают кровотечение. Различные патологии, влияющие на механические свойства фибрина, способны нарушить его функцию и привести как к образованию тромба и возможному инсульту или инфаркту, так и к повышеному кровотечению. Капсиды животных и растительных вирусов способны выдерживать колоссальное давление со стороны упакованного генетического материала. Их физические свойства, а также переходы между стабильным и нестабильным состояниями определяют жизненный цикл вирусов, в том числе созревание вируса и заражение клеток.
Современные эксперименты по изучению одиночных молекул такие как атомно-силовая микроскопия и оптические пинцеты широко используются для изучения механических свойств белковых волокон и капсул вирусов. Однако, ввиду сложности строения этих систем ( 103 105 частиц) и их больших размеров ( 50 200 нм), результаты подобных экспериментов сложно интерпретировать без понимания микромеханики исследуемых биомолекулярных систем.
Стандартные вычислительные методы молекулярной динамики (МД) в полноатомном разрешении широко используются для изучения поведения биомолекул. Но полноатомное моделирование в настоящее время ограничено размером молекулы в 10-50нм и длительностью процесса моделирования в 0, 1 10мс. Поэтому данный метод хорошо подходит только для моделирования равновесных процессов, а достижение биологически важного временного интервала от микросекунды до секунды практически невозможно даже для малых систем. В основе упрощённого метода молекулярного моделирования лежит предположение, что не все степени свободы биомолекулы одинаково важны, и за структурные единицы системы берутся не атомы системы, а их группа (например, одна аминокислота). Такой метод не только позволяет существенно сократить количество производимых вычислений, но и перейти к большему шагу по времени без потери численной стабильности системы. Графические процессоры (ГП), которые изначально были спроектированы для ускорения работы с трёхмерной графикой, способны выполнять многие вычислительные задачи, в том числе и те, которые не связаны с обработкой изображения. Программные платформы для современных ГП включают NVIDIA Compute Unied Device Architecture (CUDA) и Open Computing Language (OpenCL). CUDA, программная среда для параллельных вычислений является высокоуровневой программной платформой, расширяющей стандартные языки С и C++. Это позволяет разработчику реализовывать процедуры (ядра), которые могут выполняться одновременно во множестве независимых потоков на ГП. Комбинация упрощённого метода моделирования и высокой производительности современных ГП обладает огромным потенциалом в области молекулярного моделирования.
Ещё одним преимуществом ГП является их доступность: цена даже самых современных устройств не превышает 500 2000 долларов США, а обычный персональный компьютер, оборудованный двумячетырьмя такими устройствами, способен достигать производительности компьютерного кластера с 200 500 вычислительными ядрами.
Цели работы, объекты исследований. Объекты исследований диссертации - мономер и димер фибриногена, его фрагменты, а также капсид вируса HK97. Известные экспериментальные данные, полученные при помощи атомной силовой микроскопии, показали, что молекулы фибриногена обладают сложными механическими свойствами. Однако, эти данные не позволили точно определить источник динамических характеристик молекулы. Именно поэтому необходимо провести компьютерное молекулярное моделирование силовой денатурации фибриногена. К сожалению, ни один из существующих вычислительных методов не позволяет проводить молекулярное моделирование в условиях воздействия на молекулу, идентичных экспериментальным. Поэтому было решено адаптировать вычислительные методы для эффективной работы на ГП. Полученная реализация также была применена для объяснения динамических характеристик капсида вируса HK97.
Основной целью данной работы являлось моделирование экспериментов атомной силовой микроскопии (АСМ) на единичной молекуле. Для реализации была выбрана модель Самоорганизующегося Полимера (англ. Self Orginized Polymer, SOP). Использование модели SOP, полностью реализованной на ГП, дало возможность наблюдать микромолекулярную динамику белковых систем размером до 106 аминокислот.
Целью данной работы также являлась разработка программной реализации численных методов упрощённого моделирования белковых молекул с использованием графических процессоров. Такая комбинация позволяет моделировать большие белковые системы на экспериментальных временных интервалах без использования дорогих и сложных в обслуживании вычислительных кластеров.
Научная новизна. Данная тема является актуальной с точки зрения развития методологии комплексного исследования больших биомолекул на основе математического моделирования и вычислительных экспериментов. Представленная программная реализация для моделирования больших белковых систем на экспериментальных временных интервалах - единственная из известных на сегодняшний день. Результаты численных исследований, полученные для мономера, димера фибриногена и его фрагментов, позволили поновому интерпретировать экспериментальные данные и лучше понять микромеханику данного белка. Моделирование капсида HK показало, что динамические характеристики данной белковой системы зависят от скорости и геометрии воздействия.
Практическая ценность. Математическая модель и её численная и программная реализации были организованы как программный пакет SOP-GPU, который распространяется в виде открытого кода, сопровождается детальным руководством пользователя и не требует навыков программирования для своего использования. Таким образом, он может быть полезен другим научным группам для исследования интересующих их биомолекулярных систем.
Основные положения, выносимые на защиту:
1. Модификация математической модели самоорганизующегося полимера (SOP) и разработка численных методов для работы на графическом процессоре (ГП).
2. Реализация генераторов псевдослучайных чисел для использования в динамике Ланжевена на ГП.
3. Программный пакет SOP-GPU, размещённый в свободном доступе в сети интернет.
4. Сравнительный анализ экспериментальных данных АСМ и данных молекулярного моделирования силовой денатурации, полученных для мономера, димера фибриногена и различных фрагментов этого белка.
5. Различные динамические режимы, полученные при моделировании силовой индентации капсида вируса HK97.
Апробация работы. Полученные результаты были представлены на 238-ой и 240-ой национальных встречах американского химического общества (Вашингтон, 2009 и Бостон, 2010), на 12-ом и 13-ом конгрессах международного общества тромбоза и гемостаза (Бостон, 2009 и Киото, 2011), на научно-практической конференции “Вычисления с использованием графических процессоров в молекулярной биологии и биоинформатике” (Москва, 2010), на 53-ей научной конференции МФТИ (Долгопрудный, 2010), на конференция “Опыт и результаты исследований, проводимых под руководством приглашенных ученых-соотечественников” (Москва, 2011), на 18ой конференции “Математика. Компьютер. Образование.” (Дубна, 2011) и на 17-ой конференции по структуре и динамике биомолекул (Олбани, 2011).
Публикации. Материалы исследований опубликованы в 16-ти работах [1–16], в том числе семи [1–7] - в изданиях, рекомендованых ВАК РФ. Работы [3, 4] - в изданиях по специальности 05.13.18.
Структура и объём работы. Работа состоит из введения, шести глав, заключения, списка использованных источников, включающего 225 наименований, и трёх приложений. Диссертация изложена на 124-ёх страницах.
Основное содержание работы
Во введении обоснована актуальность темы, поставлена цель исследований, дан обзор публикаций по теме диссертации, сформулированы положения, выносимые на защиту, показаны теоретическая ценность и практическая значимость представленных в работе материалов.
В первой главе даётся краткий обзор методов молекулярного моделирования. Дан обзор основ молекулярного моделирования в явном и неявном растворителе, объяснены предпосылки использования упрощённых моделей. Приведена потенциальная функция модели самоорганизующегося полимера (Self Orginized Polymer, SOP), используемой в данной работе. Данная модель была разработана для описания механических свойств белков и моделирует процесс экспериментов на одиночных молекулах белка (таких как атомная силовая микроскопия и экспериментов при помощи оптических пинцетов). За центры взаимодействия в модели SOP берутся атомы C, а потенциальная энергия состояния белка зависит от координат {r} = r1, r2,..., rN этих атомов и задаётся выражением:
В уравнении 1, первое слагаемое описывает ковалентные связи в цепи при помощи конечно-растяжимого нелинейного эластичного потенциала (англ. Finitely Extensible Nonlinear Elastic, FENE) VF EN E.
Расстояние между соседними частицами i и j задаётся как rij, а rij - его равновесное значение (значение в нативном состоянии белка), R0 = 2 - индекс чувствительности к изменениям ковалентA ной связи. Второе слагаемое, описываемое потенциалом ЛеннардаДжонса (VN B T ), используется для описания нековалентных связей, стабилизирующих нативное состояние (нативных контактов). Значение параметра h = 0, 7 1, 6 ккал/моль характеризует силу нековалентных связей. Все прочие пары атомов (не связанные ковалентно и не формирующие нативный контакт) взаимодействуют согласно потенциалу VN B, благодаря которому становятся невозможными самопересечения цепи. Параметры r = 1.0 ккал/моль и = 3.8 постоянны и характеризуют силу и радиус отталкивания.
Основным преимуществом модели SOP является то, что несмотря на простоту силового поля, она достаточно точно описывает механическую денатурацию белков. Кроме того, благодаря использованию динамики Ланжевена в задемпфированом пределе, вычислительные процедуры остаются стабильными даже при использовании большого шага по времени в 40пс. Данная модель была успешно использована для достаточно больших систем без применения аппаратного ускорения. Однако моделирование больших систем на экспериментальных временных интервалах с использованием центрального процессора невозможно даже с применением модели SOP.
Поэтому, в данной работе модель была реализована на графических процессорах, что позволило обойти данный барьер.
Во второй главе приведен обзор публикаций по развитию графических процессоров (ГП) как альтернативных вычислительных устройств высокой производительности. Приведены примеры использования как современных ГП, так и устройств предыдущих поколений. Также во второй главе даётся обзор языка программирования CUDA, основ устройства аппаратной части и примеров его использования для научных вычислений. В последнем разделе второй главы приведены методы для оптимизации работы программы, реализованной на ГП. В биомолекулярном моделировании с использованием молекулярной динамики (МД) или динамики Ланжевена (ДЛ) взаимодействия между частицами описываются при помощи потенциальной функции (силового поля). Функциональная часть силового поля одинакова для всех частиц в системе, а уравнения движения всех частиц могут быть численно проинтегрированы независимо друг от друга. Благодаря этому, численные алгоритмы молекулярной динамики и динамики Ланжевена могут быть успешно реализованы на ГП.
В третьей главе предлагаются численные методы и алгоритмы, использованные при реализации модели SOP на ГП. Показаны два раздельных подхода к параллелизации на ГП: “Распараллеливание по частицам” и “Распараллеливание по парам взаимодействующих частиц”. Приведены процедуры создания списка Верле, численного решения уравнений движения. Далее полученная реализация была проверена на предмет аппаратных и программных ошибок. Для этого данные, полученные на графическом процессоре (ГП) были сравнены с данными, полученными на центральном процессоре (ЦП).
Результаты вычислений сравнивались также с данными, полученными аналитически. Было показано, что даже при использовании одинарной точности результаты численного эксперимента хорошо соответствуют аналитическому решению. Была проверена целесообразность использования схемы интегрирования более высокого порядка и показано, что в данном случае первого порядка точности достаточно даже при использовании шага по времени в 20-40пс. В работе впервые реализован подход “много запусков на ГП”. Данный подход позволяет полностью использовать вычислительные возможности ГП даже при моделировании сравнительно небольших систем.
В дальнейшем этот подход может быть легко расширен для использования в различных методах рассчёта термодинамики системы.
Рис. 1: Относительная производительность, или отношение скорости вычислений на ГП к скорости вычислений на ЦП (ускорение), полученное для приложения SOP-GPU как функция размера системы Ntot или количества траекторий s. Также показаны графики ускорения при использовании кеширования текстур и аппаратного ускорения математических функций. Для наглядности, график показан с логарифмическим масштабом по оси x.
Итоговая производительность разработанного приложения для ГП (программа SOP-GPU) была сравнена с оптимизированной версией реализации того же приложения для ЦП. При этом оба приложения были использованы для описания динамики Ланжевена одной и той же молекулярной системы - домена W W в состоянии равновесия (Рис. 1). Для полного задействования ресурсов ГП представленна вычислительная производительность программы SOP-GPU как функция от числа независимых траекторий, обсчитываемых одновременно на одном ГП (подход “много-траекторий-на-одном-ГП”).
Альтернативный этому вариант заключался в расчете одной траектории на одном ГП, но для систем разного размера (подход “однатраектория-на-одном-ГП”). Результаты показывают, что для небольшой системы из 34 аминокислотных остатков (домен W W ), использование одного ГП позволяет ускорить вычисления при получении трёх и более независимых траекторий, так как небольшие системы не могут полностью загрузить все параллельные вычислительные ресурсы ГП. Эквивалентную загрузку одного ГП обеспечивает запуск обсчёта одной траектории для системы из 102 частиц. В то время, как время моделирования на ЦП линейно возрастает с увеличением количества запускаемых потоков, нагрузка на ГП в таком режиме остаётся сублинейной (примерно постоянной) вплоть до 500 траекторий. Далее, ГП демонстрирует значительное превосходство в производительности над ЦП, достигая максимального ускорения в 8090 раз (Рис. 1). Для сравнения производительности были использованы ГП и ЦП одинакового технологического уровня (ГП NVIDIA GeForce GTX 295 и ЦП Quad Core Xeon 2, 66 ГГц).
В четвёртой главе приведено описание реализации генераторов псевдослучайных чисел (ГСЧ) на ГП. В силу того, что ГП не так давно стали широко использоваться в научных вычислениях, многие базовые вычислительные методы пока не реализованы и не оптимизированы для работы на этих устройствах. В этой главе рассматривается четыре различных генератора случайных чисел: линейный конгруэнтный генератор LCG, гибридный генератор “гибридный Таус”, генератор Ran2 и Фибоначчи с запаздыванием. В первой части главы даётся описание математических основ трёх методов. Далее показано, как эти методы могут быть эффективно адаптированы для работы на ГП.
Для разработки параллельных реализаций ГСЧ на ГП был использован метод разделения цикла генератора. Основная идея метода заключается в распределении одной последовательности ГСЧ, которая может быть рассмотрена как периодический цикл случайных чисел, между множеством потоков, каждый из которых производит подпоследовательность случайных чисел. Большинство ГСЧ, включая LCG, Ran2 и гибридный Таус, основаны на последовательном преобразовании текущего состояния, поэтому наиболее естественным способом получения разных последовательностей случайных чисел в разных потоках является сообщение потокам различных начальных значений состояния. При этом начальные состояния распределяются между потоками таким образом, чтобы исключить межпоточные корреляции случайных чисел. На этом основан подход “один-ГСЧ-на-поток”. Существуют ГСЧ, такие как вихрь Менсенна и алгоритм Фибоначчи, в которых можно перескочить вперёд по последовательности и вычислить случайное число (n+1), не вычисляя перед этим n-ное. Длина запаздывания, которая зависит от параметров ГСЧ, может быть выбрана больше числа потоков (которое в случае молекулярной динамики или динамики Ланжевена равно числу частиц). Тогда все N чисел могут быть получены одновременно, то есть. j-тый поток вычисляет j-тоe, (j + N )-тоe, (j + 2N )-тоe и так далее числа. В этом случае, только одно состояние ГСЧ используется для всех потоков, каждый из которых обновляет только один элемент этого состояния. Такой подход можно назвать “один-ГСЧна-все-потоки”. Реализации ГСЧ затем были проверены на предмет возникновения корреляций, также была проверена их производительность и требования к памяти.
В пятой главе описаны результаты численного моделирования силовой денатурации мономера, димера фибриногена и его фрагментов. Полученные результаты позволили по-новому интерпретировать доступные экспериментальные данные. Для исследования механизмов денатурации и для получения энергетических характеристик были использованы следующие системы: изолированный модуль, -модуль с 25-ю примыкающими аминокислотами -модуля, комплекс и -модулей (глобулярная часть домена D), фрагмент “двойной D-домен”, мономер и димер фибриногена. Для каждой системы были получены от 6 до 10 траекторий.
Экспериментальные и теоретические графики зависимости силы от растяжения для мономера, димера и олигомеров фибриногена обладали очень похожими характеристиками (Рис. 2 и 3). Статистический анализ, усреднённый по пяти траекториям моделирования, длиной 0, 2 секунды каждая, показал, что среднее значение расстояния между максимумами x 29нм для мономера фибриногена и x 27нм для димера, что хорошо соответствует значениям, полученным экспериментально. Среднее значение сил денатурации оказались равными, соответственно, f 134 и f 112пН для мономера и димера фибриногена. Эти значения несколько превосходят экспериментальные данные, что может быть следствием слишком Рис. 2: Экспериментальные результаты денатурация фибриногена при помощи АСМ. Графики зависимости силы от растяжения были получены при помощи скорости движения зонда в f = 1, 0мкм/сек для мономера фибриногена (панель (а)) и его олигомеров (панель (б)). Гистограммы распределения расстояний между пиками и сил денатурации показаны на панелях (в) и (г) соответственно. На панели (в) также показана суперпозиция трёх нормальных распределений (сплошная линия) расстояний между максимумами сил, полученных из результатов моделирования для переходов типа 1-3 (пунктирные линии), которые изображены на Рис. 3).
сильной стабилизации структуры белка в модели SOP. Главной целью моделирования было получение значений для расстояний между максимумами, значения которых хорошо соответствуют экспериментальным данным.
Основным результатом является то, что механизм денатурации фибрин(оген)а зависит от нескольких структурных элементов этой Рис. 3: Кривые зависимости силы от растяжения для мономеров (панель (а)) и димеров (панель (б)) фибриногена, полученные при помощи молекулярного моделирования с применением скорости зонда в f = 1, 0мкм/с. Точки на панелях (в) и (г) показывают расстояния между максимумами и силы денатурации, соответственно, сгруппированные для переходов типа 1-3. Максимы силы, соответствующие этим переходам, пронумерованы на графиках (панели (а) и (б).
комплексной молекулы. Главным образом, процесс ответа фибриногена на механическое воздействие состоит из обратимого процесса растяжения/сжатия свёрнутых -спиралей и последовательной денатурации глобулярных элементов -модуля. В каждом -модуле, первым событием денатурации является отделение C-терминального -луча (аминокислоты 380 392). Сразу вслед за этим, денатурирует центральная часть -модуля (аминокислоты 234 311), который при этом разделяется на два глобулярных фрагмента - Cтерминальный (аминокислоты 311 380) и N -терминальный (аминокислоты 139 234). В результате этого денатурация одного модуля происходит в три этапа, то есть в три перехода денатурации (типа 1, 2 и 3), что соответствует трём максимумам на графике зависимости силы от молекулярного растяжения.

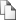
 «
«