«ПОЛНОГЕНОМНЫЙ КОМПЬЮТЕРНЫЙ АНАЛИЗ РАСПРЕДЕЛЕНИЯ САЙТОВ СВЯЗЫВАНИЯ ТРАНСКРИПЦИОННЫХ ФАКТОРОВ ЭУКАРИОТ ПО ДАННЫМ ИММУНОПРЕЦИПИТАЦИИ ХРОМАТИНА И ВЫСОКОПРОИЗВОДИТЕЛЬНОГО СЕКВЕНИРОВАНИЯ ...»
ФЕДЕРАЛЬНОЕ ГОСУДАРСТВЕННОЕ БЮДЖЕТНОЕ УЧРЕЖДЕНИЕ
НАУКИ ИНСТИТУТ ЦИТОЛОГИИ И ГЕНЕТИКИ СИБИРСКОГО
ОТДЕЛЕНИЯ РОССИЙСКОЙ АКАДЕМИИ НАУК
На правах рукописи
Орлов Юрий Львович
ПОЛНОГЕНОМНЫЙ КОМПЬЮТЕРНЫЙ АНАЛИЗ
РАСПРЕДЕЛЕНИЯ САЙТОВ СВЯЗЫВАНИЯ
ТРАНСКРИПЦИОННЫХ ФАКТОРОВ ЭУКАРИОТ ПО
ДАННЫМ ИММУНОПРЕЦИПИТАЦИИ ХРОМАТИНА И
ВЫСОКОПРОИЗВОДИТЕЛЬНОГО СЕКВЕНИРОВАНИЯ
03.01.09 – математическая биология, биоинформатика Диссертация на соискание ученой степени доктора биологических наук
Научный консультант:
академик РАН, д.б.н. Н.А. Колчанов Новосибирск -
ОГЛАВЛЕНИЕ
ВВЕДЕНИЕ Список сокращений Глава 1. ОБЗОР ЛИТЕРАТУРЫ1.1. ЗАДАЧИ КОМПЬЮТЕРНОГО АНАЛИЗА ГЕНОМНЫХ ДАННЫХ
1.1.1. Международные проекты геномных исследований 1.1.2. Статистические методы и алгоритмы 1.2 ТРАНСКРИПЦИЯ ГЕНОВ ЭУКАРИОТ 1.2.1. Транскрипция и транскрипционные факторы 1.2.2. Методы измерения экспрессии генов1.3 РЕГУЛЯТОРНЫЕ УЧАСТКИ ГЕНОВ: ПРОМОТОРЫ И ЭНХАНСЕРЫ
1.3.1. Промоторы и энхансеры 1.3.2. Компьютерные методы распознавания регуляторных районов генов 1.3.3. Предсказание сайтов связывания нуклеосом 1.3.4. Полногеномные методы определения сайтов связывания транскрипционных факторов ChIP-seq и ChIP-PET 1.3.5. Задачи исследования распределения сайтов связывания транскрипционных факторов в геноме по данным ChIP-seq 1.4. ТРАНСКРИПЦИОННЫЕ ФАКТОРЫ – ОНКОГЕНЫ И ПРОБЛЕМЫИССЛЕДОВАНИЯ ИХ РЕГУЛЯЦИИ
1.4.1. Транскрипционные факторы p53, STAT1, FOXA1 1.4.2. Транскрипционный фактор c-Myc 1.4.3. Транскрипционный фактор рецептор эстрогенов 1.4.4. Возникновение опухолей и регуляция транскрипции 1.4.5. Задачи анализа регуляции транскрипции онкогенов 1.5. ФАКТОРЫ ПОДДЕРЖАНИЯ ПЛЮРИПОТЕНТНОСТИ ВЭМБРИОНАЛЬНЫХ СТВОЛОВЫХ КЛЕТКАХ
1.5.1. Эмбриональные стволовые клетки 1.5.2. Транскрипционные факторы плюрипотентности и репрограммирование 1.5.3. Эффективность репрограммирования и дополнительные факторы 1.5.4. Задачи по определению сайтов связывания факторов в ЭСК1.6. ПРОСТРАНСТВЕННЫЕ КОНТАКТЫ ХРОМОСОМ В ЯДРЕ
1.6.1. Проблема исследования контактирующих участков хромосом 1.6.2. Методы определения хромосомных контактов с помощью секвенирования: 3С и Hi-C 1.6.3. Метод ChIA-PET 1.6.4. Постановка задач анализа данных ChIA-PETЗАКЛЮЧЕНИЕ ПО ОБЗОРУ ЛИТЕРАТУРЫ И ПОСТАНОВКА ЗАДАЧ
ИССЛЕДОВАНИЯ
ПЛАН И СТРУКТУРА ИССЛЕДОВАНИЯ
Глава 2. МОДЕЛИ РАСПРЕДЕЛЕНИЯ САЙТОВ СВЯЗЫВАНИЯ ВГЕНОМЕ
2.1 Введение. Компьютерные модели и базы данных 2.2 Компьютерная обработка данных ChIP-seq 2.2.1. Компьютерный анализ профиля связывания ChIP-seq в геноме и статистическое определение пиков 2.2.2. Определение статистической значимости найденных пиков профиля связывания ChIP-seq 2.2.3. Фильтрация профиля связывания ChIP-seq по геномной аннотации 2.3. Метод оценки полноты (сатурации) эксперимента ChIP-seq 2.4. Определение генов-мишеней транскрипционных факторов по данным экспрессии генов на микрочипах 2.5 Оценка качества сигнала экспрессии на микрочипах Affymetrix 2.6. База данных RatDNA специализированных микрочипов генов крысы 2.7. Модели регуляторных районов транскрипции включающие антисенс транскрипты 3.2. Распределение сайтов связывания транскрипционного фактора c-Myc, определенное по методу ChIP-PET 3.3. Исследование распределения сайтов связывания ТФ рецептора эстрогенов ER с помощью ChIP-seq 3.4. Распределение сайтов связывания транскрипционных факторов плюрипотентности по данным ChIP-seq 3.5 Регуляторные контуры взаимодействий генной сети по данным связывания транскрипционных факторов 3.6 Энхансеры и множественные локусы регуляции транскрипции по данным ChIP-seq 3.7 Компьютерное исследование ко-локализации в геноме и построение тепловых карт кластеров сайтов связывания 3.8. Дальнейшие исследования ССТФ в ЭСК мыши с помощью ChIP-seq 3.10. Сайты связывания в геноме в зависимости от дозового эффекта и взаимодействия ко-факторов на примере ССТФ Smad2 в ЭСК мыши 3.11. Геномные карты сайтов связывания ТФ для генома человекаТРАНСКРИПЦИОННЫХ ФАКТОРОВ В ГЕНОМЕ
4.2. Исследование нуклеосомной упаковки и расположения сайтов связывания транскрипционных факторов в геноме дрожжей 4.2. Исследование позиционирования нуклеосом и эффективности трансляции генов у дрожжей 4.2. Исследование ассоциации сайтов связывания ТФ с модификациями хроматина 4.4 Предсказание сайтов связывания в геноме человека с помощью компьютерной модели, учитывающей состояние хроматина 4.5. Общая зависимость доступности ССТФ от состояния хроматина опосредована присутствием нуклеосом на ДНК 4.6. Заключение к Главе. Общая проблема предсказания сайтов связывания на основе данных о модификациях хроматинаТРАНСКРИПЦИИ В ГЕНОМЕ ЧЕЛОВЕКА
5.1. Введение к Главе 5. Проблема исследования хромосомных контактов 5.1. Принципы построения карт хромосомных взаимодействий и компьютерные модели 5.2. Анализ трехмерной структуры генома через секвенирование. ChIA-PET, Hi-C технологии 5.3 Хромосомные контакты, опосредованные связыванием транскрипционного фактора ER в геноме человека 5.4. Хромосомные контакты, опосредованные комплексом РНК-полимеразы II в геноме человекаЗАКЛЮЧЕНИЕ И ОБСУЖДЕНИЕ
ВЫВОДЫ ПО ДИССЕРТАЦИОННОЙ РАБОТЕ
Список публикаций по теме диссертации Список литературыПРИЛОЖЕНИЕ
ВВЕДЕНИЕ
Актуальность проблемы Начало XXI века ознаменовано значительными достижениями в молекулярной биологии и генетике, связанными с качественно новыми, полногеномными исследованиями. Создание высокопроизводительных автоматизированных систем секвенирования ДНК позволяет эффективно секвенировать (расшифровывать) протяженные последовательности ДНК, вплоть до целых геномов [1, 2]. Выполняются крупномасштабные проекты полного секвенирования геномов эукариот, что ведет к лавинообразному росту объема информации как о полных последовательностях геномов эукариот (http://www.ncbi.nlm.nih.gov/genbank/statistics), так и о последовательностях регуляторных районов экспрессии генов. Качественный скачок в развитии технологий массового параллельного секвенирования, таких как Roche 454, Illumina Solexa, SOLiD, за последние 5-10 лет дал импульс серии новых исследований в молекулярной биологии [2-5]. Продолжаются проекты по исследованию генетического разнообразия, полиморфизмов в популяциях [6, 7], - в настоящее время доступно более тысячи полностью секвенированных индивидуальных геномов человека. В основных молекулярно-генетических банках данных (EMBL, GenBank, DDBJ) накоплена информация о более чем 20 тысячах полностью секвенированных геномах микроорганизмов и полутора тысячах геномов эукариот, включая геном человека, причем объем расшифрованных последовательностей стремительно растет. Разработка методов высокого разрешения для анализа особенностей организации регуляторных районов генов и структуры хроматина в масштабе генома дает качественно новые данные для исследования молекулярных механизмов регуляции транскрипции генов и ставит новые задачи перед компьютерной геномикой и биоинформатикой, в том числе в проекте ENCODE [8].В последние годы благодаря методам высокопроизводительного секвенирования ChIP-seq, ChIP-on-chip, ChIP-PET и другим ChIP-технологиям, сопряженным с иммунопреципитацией хроматина (ChIP - Chromatin ImmunoPrecipitation), появился огромный массив качественно новых данных, позволяющих оценить регуляторный потенциал клетки, в том числе исследовать все сайты связывания заданного транскрипционного фактора в геноме [9-12].
Представляемая диссертационная работа посвящена применению современных математических и компьютерных методов анализа регуляции транскрипции эукариот с использованием данных ChIP-экспериментов, связанных с секвенированием и иммунопреципитацией хроматина.
Исследование регуляции экспрессии генов эукариот в масштабе генома требует изучения сайтов связывания транскрипционных факторов (СТТФ), контролирующих транскрипцию генов, их геномной локализации, определения генов-мишеней ТФ.
Оценка числа сайтов связывания, предсказанных по нуклеотидной последовательности, только для одного транскрипционного фактора в геноме человека может достигать миллиона сайтов, что значительно превышает число генов в геноме [13]. В то же время, экспериментально установленное число сайтов варьирует от нескольких тысяч до десятков тысяч, превышая число потенциальных генов-мишеней. При этом большая часть сайтов связывания располагается в удаленных от генов районах, дистальных энхансерах, что затрудняет их компьютерное предсказание и экспериментальное исследование [3].
В последние десятилетия использовались такие подходы к определению сайтов связывания регуляторных белков, как футпринтинг ДНК, методы задержки пробы в геле (ретардация). Однако этими методами невозможно исследовать все сайты связывания транскрипционного фактора (ССТФ) в геноме. Прямое применение таких экспериментальных методов для поиска, сравнения, картирования огромного числа всех сайтов связывания, описания регуляторных районов генов в геноме невозможно из-за их большой трудоемкости и значительной стоимости.
Встают задачи исследования механизмов регуляции экспрессии генов на уровне транскрипции, связанные с развитием высокоэффективных экспериментальных методик измерения экспрессии генов, изучения динамических профилей транскрипции построения карт ДНК-белковых и регуляторных взаимодействий Существующие микрочиповые технологии позволяют изучать динамику экспрессии тысяч генов одновременно [14]. Систематизация и анализ этих огромных объемов экспериментальных данных геномики и транскриптомики является сложнейшей задачей, связанной как с фундаментальными вопросами биоинформатики и системной биологии, так и с биотехнологическими приложениями, медициной, фармацевтикой.
Методы иммунопреципитации хроматина (ChIP-on-chip, ChIP-PET, ChIP-seq) с последующим массовым параллельным секвенированием позволяют исследовать сайты связывания транскрипционных факторов в масштабе генома, ставя новые задачи биоинформатики для адекватной идентификации сайтов [9, 15-18]. Исследование структуры хроматина на уровне отдельных нуклеосом (модификаций метилирования и ацетилирования гистонов в определенных позициях) с помощью технологий ChIP-seq качественно дополняет описание регуляторных районов генов в масштабе генома [13, 19, 20]. Важным направлением исследования является построение полногеномных карт известных регуляторов плюрипотентности NANOG, OCT4, SOX2, KLF4 в стволовых клетках человека и мыши. Использование иммунопреципитации хроматина позволяет опосредованные белковыми комплексами [21-23]. Накопилось большое количество экспериментальных данных о роли трехмерной организации генома в регуляции экспрессии генов (удаленные энхансеры, пространственные домены), полученных с помощью технологий секвенирования. Недавно появившиеся методы исследования трехмерных хромосомных контактов Hi-C [24] и ChIA-PET [12] дают качественно новую информацию о регуляторных последовательностях в геноме.
компьютерах стали незаменимым инструментом в экспериментальной работе молекулярных биологов. За последние десятилетия был создан широкий круг последовательностей сайтов связывания, представления их в форме весовых матриц, скрытых марковских моделей, и последующего распознавания сайтов в протяженных последовательностях [27], что дает основу для теоретического компьютерного описания регуляторных районов. Большинство алгоритмов, заложенных в эти программы, применяют технику теории вероятностей и математической статистики [29], дискретной математики [30] для исследования статистических свойств и закономерностей в строении последовательностей биополимеров [27, 28]. Обработка высокопроизводительных вычислительных кластеров [28].
Важнейшей проблемой биоинформатики является проблема компьютерного исследования и поиска в геноме последовательностей, регулирующих экспрессию генов эукариот. Если раньше, в 1990-е годы, объектом исследования были одиночные последовательности и выборки последовательностей, небольшие компиляции данных и базы данных, отдельные хромосомы, и, соответственно, задачи анализа были ограничены имеющимся на тот момент объемом данных [31], то сейчас ставится задача полногеномного анализа с использованием гетерогенных интегрированных информационных ресурсов, касающихся различных аспектов организации геномов [8, 28]. К таким ресурсам, содержащим полногеномные данные, относятся базы данных экспрессии генов на микрочипах - Gene Expression Atlas [32], BioGPS [33], репозитарии экспериментов секвенирования - GEO NCBI [34]), интегрированные средства хранения данных и визуализации геномной информации - Ensembl [35], UCSC Genome Browser [36].
Одной из ключевых задач является полногеномный компьютерный анализ распределения сайтов связывания транскрипционных факторов в геноме человека и в модельных генах эукариот по данным иммунопреципитации хроматина и высокопроизводительного секвенирования, что ставит новые задачи перед биоинформатикой, представленные в настоящей работе.
Цель и задачи исследования Цель работы – компьютерная реконструкция структуры регуляторных районов, контролирующих транскрипцию генов эукариот на основе анализа данных о положении сайтов связывания транскрипционных факторов в геноме, полученных с помощью технологии иммунопреципитации хроматина и высокопроизводительного секвенирования (ChIP-seq).
Для достижения этой цели решались следующие задачи:
1. Разработка методов анализа данных секвенирования ChIP-seq и создание статистической модели полногеномного распределения сайтов связывания транскрипционных факторов (ССТФ).
2. Компьютерная реконструкция полногеномных карт сайтов связывания транскрипционных факторов плюрипотентности c-Myc, Oct4, Nanog, Sox2, E2f1, n-Myc, Tbx3, Eset, Nr5a2, Smad2 в геноме мыши. Реконструкция распределения сайтов связывания транскрипционных факторов MYC, PRDM14, ER, FOXA1, OCT4, NANOG в геноме человека.
транскрипционного фактора ER с определенными с помощью технологии ChIP-seq маркерами хроматина, в частности, модификациями гистона H3 (H3K4me3, H3K4me1, H3K27me3, H3K9me3, H3K9ac, H3K14ac), и создание метода предсказания сайтов связывания транскрипционного фактора ER в геноме человека на основе профилей модификаций гистонов.
4. Изучение роли хромосомных контактов в регуляции транскрипции генов человека на моделях РНК-полимеразы II и транскрипционного фактора ER на основе компьютерного анализа полногеномных данных ChIP-seq и ChIA-PET.
Методические задачи, решавшиеся в диссертации, включали: разработку и компьютерную реализацию на языках С++ и R: (1) алгоритмов анализа полногеномных профилей связывания транскрипционных факторов ChIP-seq; (2) алгоритмов анализа нуклеотидных последовательностей регуляторных районов, формируемых ССТФ;
(3) алгоритма анализа полноты эксперимента ChIP-seq и ChIP-PET; (4) алгоритма определения кластеров ССТФ в геноме: (5) программ обработки данных экспрессии генов на микрочипах; (6) программ интеграции данных геномной аннотации расположения генов и профилей ChIP-seq; (7) программ анализа профилей ChIA-PET и ChIP-seq.
В качестве экспериментальной информации, которая была проанализирована с помощью компьютерных методов, разработанных автором диссертации, использовались данные, полученные соавторами научных публикаций Ng H.H., Kong S.
Joseph R., Liu E.T., Ruan Y., Wei C.L., Lee K.L., Clarke N. с помощью методов секвенирования ДНК в Геномном институте Сингапура, а также публично доступные данные секвенирования из GEO NCBI. Автор диссертации выражает своим коллегам благодарность за предоставление этих данных.
Научная новизна Разработаны оригинальные программы анализа распределения сайтов связывания транскрипционных факторов в геноме на основе анализа данных секвенирования сопряженного с иммунопреципитацией хроматина ChIP-seq [16, 37, 38]. С помощью этих программ построены карты связывания транскрипционных факторов c-Myc, Oct4, Nanog, Sox2, E2f1, n-Myc, Tbx3, Eset, Nr5a2, Smad2 в геноме мыши [3, 39-41], транскрипционных факторов c-Myc [9], ER [13], PRDM14 в геноме человека [42], Zic в геноме рыбы D.rerio [43]. Все карты ССТФ были получены впервые.
Компьютерные программы интеграции данных о геномной локализации указанных выше ССТФ и уровнях экспрессии генов, измеренных с помощью микрочипов, позволили получить новые данные о регуляции транскрипции генов [3, 37, 44, 45]. Разработанная компьютерная база данных наборов проб микрочипов платформы Affymetrix U133, включающая оценки качества проб – однозначности картирования, соответствия целевым (таргетным) транскриптам, была новой на момент публикации, и использовалась для анализа присутствии транскриптов в цис-антисенс ориентации [46-49].
Исследование распределения нуклеосом в промоторных районах с помощью собственных компьютерных программ и анализа данных прямого секвенирования нуклеосомной ДНК дрожжей [50, 51] показало отсутствие предпочтения к позиционированию нуклеосом in vivo по сравнению с данными in vitro и теоретическим предсказанием на основе контекста.
Компьютерный анализ впервые показал статистически значимую совместную локализацию сайтов связывания транскрипционных факторов Oct4, Sox2, Nanog, с одной стороны и c-Myc, n-Myc с другой, в эмбриональных стволовых клетках (ЭСК) мыши, рассчитанную по данным ChIP-seq [3, 40, 41]. Объединенные полногеномные карты расположения сайтов связывания транскрипционных факторов в геноме человека для эмбриональных стволовых клеток (ЭСК) впервые представлены в форме матриц сближенности (тепловых карт) [3, 52]. Впервые получено распределение сайтов связывания транскрипционного фактора PRDM14 в геноме для ЭСК человека и определен нуклеотидный мотив связывания [42].
Впервые построен компьютерный метод предсказания сайтов связывания ТФ ER в масштабе генома на основе профилей модификации хроматина - ацетилирования и метилирования гистона H3 (H3K4me3, H3K4me1, H3K27me3, H3K9me3, H3K9ac, H3K14ac), определенных с помощью технологии ChIP-seq в клеточных линиях MCF- и T47D [13, 37]. Данные по модификациям хроматина для 16 библиотек ChIP-seq в первый раз использовались в едином компьютерном исследовании для компьютерного предсказания связывания ER.
С помощью разработанных автором компьютерных программ карты хромосомных контактов, опосредованных связыванием белка рецептора эстрогенов ER [21], полученные посредством технологии секвенирования парных концов ChIAPET в клетках MCF-7, впервые проанализированы совместно с данными ChIP-seq.
Впервые на основе компьютерного анализа интегрированных полногеномных данных о хромосомных контактах, опосредованных комплексами РНК-полимеразы II, сайтах связывания транскрипционных факторов, транскрипционной активности генов, и профилей модификаций гистонов для пяти клеточных линий в геноме человека показана положительная корреляция участков хромосомных контактов с модификациями гистонов, характеризующими открытое состояние хроматина (H3K4me3, H3K9ac, H3K4me1) [12].
Теоретическое значение работы. Разработанная компьютерная статистическая модель распределения сайтов связывания транскрипционных факторов позволяет достоверно определять локализацию ССТФ в геноме и оценивать полноту эксперимента по координатам секвенированных прочтений ChIP-seq.
предсказания локализации сайтов связывания транскрипционного фактора - рецептора эстрогенов ER в геноме человека за счет одновременного анализа как нуклеотидных последовательностей, так и профилей модификации хроматина (ацетилирования и метилирования гистонов), рассчитанных по данным ChIP-seq.
Представлена компьютерная модель хромосомных петель регуляторных районов транскрипции в геноме человека, опосредованных комплексом РНК-полимеразы II основанная на данных ChIA-PET.
Научно-практическая ценность разработанных методов состоит в программах анализа регуляторных районов генов по данным секвенирования в масштабе генома, полученных картах сайтов связывания сайтов связывания транскрипционных факторов Oct4, Nanog, Sox2, E2f1, n-Myc, c-Myc, Tbx3, Eset, Nr5a2, Smad2 в геноме мыши, онкогенов MYC и ER в геноме человека.
Программный комплекс ICGenomics (http://www-bionet.sscc.ru/icgenomics/) для исследования регуляторных районов генов и функциональной аннотации геномных последовательностей обеспечивает существенное расширение методов компьютерного анализа полногеномных данных [44]. Разработана база данных цис-антисенс транскриптов и качества проб микрочипов Affymetrix U133 [46, 47], база данных экспрессии генов на микрочипах для крыс RatDNA [53] (свидетельство госрегистрации базы данных RatDNA № 621051 от 10.10.2012г.).
Созданное Интернет-доступное программное обеспечение позволяет выполнять анализ распределения сайтов связывания транскрипционных факторов, их функциональную аннотацию (http://www-bionet.sscc.ru/icgenomics/, http://pixie.bionet.nsc.ru/ratdna/rat/index.php).
Разработан учебный курс по компьютерной геномике (Кафедра информационной биологии ФЕН НГУ), учебные материалы представлены на Школе молодых ученых по системной биологии и биоинформатике SBB-2013 (http://conf.nsc.ru/sbb2013), съездеконференции ВОГиС-2013.
По тематике данной работы выполнены госконтракты Министерства образования и науки РФ на разработку программного обеспечения для геномных исследований (№07.514.11.4003 «Разработка алгоритмов и программных систем для решения задач анализа последовательностей, возникающих в теоретической и прикладной геномике», № 16.513.12.3107 «Проведение проблемно-ориентированных поисковых исследований в области ДНК-чипов в рамках технологической платформы «Медицина будущего»», № 16.512.11.2274 «Проведение проблемно-ориентированных поисковых исследований по тематике технологической платформы "Медицина будущего" в области поиска молекулярных мишеней онкологических заболеваний с помощью биоинформационных и постгеномных технологий»), гранты РФФИ (00-04-49229-а, 01-07-90376-в, 02-07в, 03-04-48506-а, 03-04-48555-а, 03-07-90181-в, 03-07-96833-р2003югра_в, 05-04- 49111-а, 05-07-90185-в, 05-07-98012-р_обь_в, 11-04-01771-а, 11-04-01888-а, 11-04- 92712-ИНД_а, 12-04-00897-а, 14-04-01906), Интеграционные проекты СО РАН (119), проект 8740 Минобрнауки России «Научные и научно-педагогические кадры инновационной России» на 2009 – 2013 годы «Интегрированная биоинформационная платформа анализа данных экспрессии генов в тканях мозга», начата работа по гранту РНФ 14-14-00269.
Положения, выносимые на защиту 1) Разработанная статистическая модель полногеномного распределения сайтов связывания транскрипционного фактора позволяет оценивать полноту эксперимента по секвенированию и иммунопреципитации хроматина ChIP-seq и рассчитывать статистически значимые оценки нижней и верхней границ общего числа сайтов связывания в геноме для исследуемого фактора.
2) Полногеномные карты сайтов связывания транскрипционных факторов в эмбриональных стволовых клетках, построенные по данным ChIP-seq для c-Myc, Oct4, Nanog, Sox2, E2f1, n-Myc, Tbx3, Eset, Nr5a2, Smad2 в геноме мыши свидетельствуют о совместной локализации групп сайтов связывания транскрипционных факторов Oct4, Sox2, Nanog, с одной стороны, и c-Myc, n-Myc с другой.
3) Нуклеотидные последовательности, окружающие сайты связывания транскрипционного фактора Smad2 в геноме мыши, содержат специфические группы нуклеотидных мотивов, соответствующих потенциальным сайтам связывания других транскрипционных факторов. Эти мотивы различаются для сайтов связывания Smad2, найденных в эмбриональных стволовых клетках мыши при действии внешних факторов - белка Activin и ингибитора SB431542, соответственно.
4) Расположение сайтов связывания транскрипционного фактора ER в геноме человека положительно ассоциировано с районами метилирования и ацетилирования гистонов нуклеосом H3K4me3, H3K4me1, H3K9ac и H3K14ac. Разработан компьютерный алгоритм для предсказания сайтов связывания ER в геноме по ChIPseq маркерам состояния хроматина; показана высокая точность предсказания с помощью этой модели.
5) Геномные области хромосомных контактов, опосредованных комплексом РНКполимеразы II, обогащены сайтами связывания транскрипционных факторов и участками модификаций гистонов, связанными с активацией экспрессии генов.
Личный вклад автора.
Все представленные в диссертации результаты получены лично автором.
Публикации, представленные в данной работе, были написаны в соавторстве. Роль автора в статьях, где он не являлся первым автором или автором для переписки, обозначена как «analyzed the data» (компьютерный и теоретический анализ данных, статистическая обработка). Специально для данного исследования автором были написаны компьютерные программы анализа ChIP-seq, статистического моделирования, сравнения геномных координат и геномной аннотации, оценки качества наборов проб микрочипов, анализа нуклеотидных контекстов, генерации базы данных цис-антисенс транскриптов, компьютерной симуляции полногеномных данных ChIP-PET, ChIP-seq и ChIA-PET.
Ключевые биоинформатические разработки по анализу наборов проб экспрессионных микрочипов Affymetrix U133 представлены в публикациях, где автор диссертации является первым автором статей (Orlov et al. 2007a; Orlov et al., 2007b;
Орлов и соавт., 2011) [46, 47, 49]. База данных цис-антисенс транскриптов в геноме человека, интегрированная с расположением наборов проб Affymetrix U133, опубликована в статье (Grinchuk,... Orlov et al., 2010) [48]. Методика анализа полноты эксперимента ChIP-seq представлена в работе (Orlov et al., 2009) [38]. Компьютерные программы, разработанные автором, и их применения описаны в работах (Orlov et al., 2012; Kuznetsov, Orlov et al., 2007; Орлов и соавт. 2012; Орлов, 2014) [16, 37, 44, 55].
Применения к анализу экспрессии мобильных элементов представлены в работе (Орлов и соавт., 2011) [49].
В статье (Joseph, Orlov et al., 2010) [13] посвященной исследованию сайтов связывания транскрипционного фактора ER в геноме человека автор диссертации имеет равное первое авторство.
Основные результаты применения разработанных компьютерных методов для анализа распределений сайтов связывания транскрипционных факторов представлены в статьях, написанных в соавторстве. В статье (Chen, … Orlov et al., 2008) [3] автор диссертации выполнил компьютерный анализ данных и оценил полноту эксперимента ChIP-seq для 13 различных транскрипционных факторов. Программа анализа профиля ChIP-seq, разработанная автором, использовалась в исследованиях транскрипционных факторов плюрипотентности для ЭСК мыши, опубликованных в статьях [39-41] (Yuan, … Orlov et al., 2009; Heng, … Orlov et al., 2010; Han, … Orlov et al., 2010), а также (Lee, … Orlov et al., 2011) [54].
В статье (Chia, … Orlov et al., 2010) [42] автором диссертации выполнен анализ реконструкция генных сетей плюрипотентности в ЭСК человека. В статье (Zhao, … Orlov et al., 2007) [19] с помощью разработанных компьютерных моделей исследованы полногеномные данные модификаций гистонов H3K4me3 и H3K27me3. Обобщение анализа распределений ССТФ в ЭСК человека и мыши дано в статье (Heng, Orlov, Ng, 2010) [52]. В статье (Winata, …Orlov et al., 2013) [43] представлено применение разработанных автором программ для анализа расположения ССТФ в геноме D.rerio, впервые выполненном на данных ChIP-seq для этого организма.
Публикация (Zeller, … Orlov et al., 2006) [9] содержит описание применения компьютерных моделей ССТФ анализа эксперимента ChIP-PET для ТФ c-Myc. В работе (Goh, Orlov et al., 2010) [51] с помощью разработанной автором компьютерной программы исследован профиль плотности нуклеосом в геноме дрожжей. В статьях (Fullwood, … Orlov et al., 2009) [21] и (Li, …Orlov et al., 2012) [12] вклад автора состоял в компьютерной обработке данных хромосомных контактов, полученных методом ChIA-PET.
Работы автора, приведенные в списке литературы и не перечисленные выше, носили методический характер, и относились к применениям разработанных алгоритмов (Орлов и соавт. 2006; Воробьева, …, Орлов и соавт. 2005; Orlov et al.. 2006;
Guo, … Orlov et al., 2010; Путта, Орлов и соавт., 2011; Суслов, …. Орлов, 2013) [50, 56исследованию экспрессии генов на микрочипах (Кожевникова, … Орлов, 2012;
Kozhevnikova,… Orlov et al. 2013; Медведева, … Орлов, 2013) [45, 53, 60].
Апробация работы. Результаты были представлены на Пятой, Шестой, Седьмой, Восьмой и Девятой Международных Конференциях по Биоинформатике и Регуляции Структуры Генома (BGRS'06, BGRS'08, BGRS\SB-2010, BGRS\SB-2012 и BGRS\SBНовосибирск, 2006, 2008, 2010, 2012 и 2014 гг.), конференциях HUGO (2008, Хайдарабад, Индия; 2010, Монпелье, Франция; 2013, Сингапур), конференции-школе CSHL-UK – 2007 (Хинкстон, Великобритания), Конференции A-STAR 2010г.
(Сингапур), Международном Симпозиуме по Биотехнологии (Москва, 2011), Школе по биоинформатике BREW-2011 (Тарту, Эстония), конференциях Постгеном- (Новосибирск) и Постгеном-2012 (Казань), Конференции по интегративной Биоинформатике IB-2012 (Ханчжоу, Китай), Международном Семинаре по Системной биологии и медицине SysPatho-2012 (Санкт-Петербург), конференциях ВОГиС- (Новосибирск), MCCMB-2013 (Москва), «Нейроинформатика-2014» (Москва).
Публикации. По теме диссертации опубликовано 33 печатные работы, из них – статьи в научных изданиях (журналы по списку ВАК). Включая тезисы конференций, общее число публикаций по теме диссертации - 52.
Структура и объем работы Диссертация состоит из пяти глав: «Обзор литературы», «Модели распределения сайтов связывания транскрипционных факторов в геноме», «Карты сайтов связывания по данным ChIP-seq», «Модификации хроматина и связывание транскрипционных факторов по данным ChIP-seq», «Хромосомные контакты и регуляция транскрипции в геноме человека». Вторая глава описывает разработку методов компьютерного анализа данных ChIP-seq и анализа экспрессии генов. Третья, четвертая и пятая главы описывают применение разработанных средств для анализа ССТФ в ЭСК человека и мыши, построение полногеномных карт, анализ распределения сайтов связывания рецептора эстрогенов ER. В четвертой главе анализ ССТФ рассмотрен в контексте структуры хроматина и модификаций гистонов, в пятой – с точки зрения хромосомных контактов. Объем диссертации составляет 343 машинописных страницы, включая рисунков и 28 таблиц. Список литературы содержит 521 ссылку.
Обзор литературы (Глава 1) содержит информацию о современных исследованиях регуляторных районах транскрипции в геноме человека, включая определение сайтов связывания с помощью технологий иммунопреципитации хроматина (ChIP).
Представлены исследования по регуляции экспрессии генов, связанных с образованием опухолей (ESR1, MYC, TP53), тканеспецифичной экспрессии в клеточных культурах.
Описаны подходы к изучению эмбриональных стволовых клеток (ЭСК) человека и мыши, показана роль транскрипционных факторов плюрипотентности в репрограммировании. Дан обзор проблем исследования трехмерных контактов хромосом в ядре с помощью секвенирования (методы 3C, Hi-C и ChIA-PET).
Рисунок 1.1 представляет логическую взаимосвязь Глав диссертационной работы, потоков данных и видов выполненного компьютерного анализа. Из схемы, представленной на рисунке, видно, что Глава 2 «Модели распределения сайтов связывания транскрипционных факторов в геноме» представляет компьютерные модели и алгоритмы, применение которых для полногеномного анализа сайтов связывания транскрипционных факторов (ССТФ) по данным ChIP-seq описано в следующей главе «Карты сайтов связывания по данным ChIP-seq».
Глава «Обзор литературы».
Постановка задач исследования Глава «Модели распределения сайтов связывания Компьютерные алгоритмы анализа профилей ChIP-PET и ChIP-seq, Глава «Карты сайтов связывания по данным ChIP-seq»
Применения компьютерных алгоритмов для построения и анализа карт Глава «Модификации хроматина и Глава «Хромосомные контакты и связывание транскрипционных регуляция транскрипции в геноме Компьютерный анализ данных ChIP-seq Компьютерная интеграция данных ChIA-PET о нуклеосомной упаковке, модификациях о хромосомных контактах и данных ChIP-seq Приложение. Коды компьютерных программ. Построенные Рис. 1.1. Взаимосвязь глав диссертационной работы.
Как показано на рисунке, дальнейшее применение анализа данных ChIP-seq представлено в Главах «Модификации хроматина и связывание транскрипционных факторов по данным ChIP-seq» и «Хромосомные контакты и регуляция транскрипции в геноме человека», которые также связаны между собой объектом исследования данными о ССТФ и модификациях гистонов в геноме человека.
Глава 2 «Модели распределения сайтов связывания транскрипционных факторов в геноме» содержит описание разработанных методов и компьютерных моделей распределения сайтов связывания транскрипционных факторов в эукариотическом геноме на основе анализа профилей ChIP-seq. Представлены алгоритмы анализа данных ChIP-seq о связывании транскрипционных факторов в геноме и базы микрочиповых данных по экспрессии генов, разработанные автором [3, 9, 13, 16, 38].
Представлены модели регуляторных районов транскрипции, включающие антисенс транскрипты, описан анализ качества наборов проб микрочипа Affymetrix U133 [46, 47, 49], построение базы данных цис-антисенс транскриптов [48, 61]. Показаны примеры применения компьютерного анализа экспрессии генов на микрочипах для генов крысы [45, 53]. Описаны общие средства компьютерной интеграции геномных данных, разработанные в ИЦиГ СО РАН [49, 50, 57-60], включая программный комплекс ICGenomics [44].
Глава 3 «Карты сайтов связывания по данным ChIP-seq» посвящена описанию карт сайтов связывания транскрипционных факторов построенных автором по экспериментальным данным ChIP-seq в геноме человека, в геноме мыши и в геноме D.rerio [9, 13, 39, 41-43, 54]. С помощью разработанных компьютерных программ обработки данных ChIP-PET и ChIP-seq были проанализированы исходные данные и определены сайты связывания транскрипционных факторов c-Myc, STAT1, FOXA1, ER, PRDM14 [9, 13, 42] в геноме человека, а также сайты связывания транскрипционных факторов Nanog, Oct4, Sox2, Klf4, E2f1, Esrrb, CTCF, n-Myc, c-Myc, Smad1, STAT3, Tcfcp21l, Zfx, Suz12 в геноме мыши [3]. Исследовано распределение ССТФ генов, ответственных за поддержание плюрипотентности в эмбриональных стволовых клетках (ЭСК) мыши; показано существование кластеров сайтов связывания факторов Oct4-Nanog-Sox2 [3]. Представлены аналогичные кластеры связывания OCT4NANOG-SOX2 в ЭСК в геноме человека [42, 52].
Глава 4 «Модификации хроматина и связывание транскрипционных факторов по данным ChIP-seq» содержит описание применения разработанных компьютерных методов к исследованию модификаций хроматина и связыванию транскрипционных факторов в геноме дрожжей [51, 62, 63] и в геноме человека [19]. Проанализированы полногеномные данные по модификациям гистонов (ацетилирования и метилирования гистона H3) и сайтам связывания транскрипционных факторов ER, FOXA1 в геноме человека [13, 21, 37]. Представлен компьютерный метод предсказания сайтов связывания ER в масштабе генома на основе профилей модификаций гистонов (H3K4me3, H3K4me1, H3K27me3, H3K9me3, H3K9ac, H3K14ac), определенных с помощью технологии ChIP-seq. Представлено обсуждение результатов в связи с продолжающимися геномными исследованиями [12].
Глава 5 «Хромосомные контакты и регуляция транскрипции в геноме человека»
представляет исследование хромосомных контактов, полученных с помощью массового параллельного секвенирования нуклеотидных последовательностей контактирующих участков хромосом по методу ChIA-PET для ER и комплекса РНКполимеразы II в геноме человека, с помощью разработанных автором диссертации компьютерных программ [12, 21, 64]. Показана ассоциация участков хромосомных контактов с регуляторными районами транскрипции генов и модификациями хроматина в геноме человека [12].
В Приложении даны коды программ и схемы алгоритмов, таблицы, содержащие координаты сайтов в геноме, результаты анализа кластеризации ССТФ, описание использованных компьютерных ресурсов.
Научно-практическая ценность Практическое применение методов анализа функциональных участков (ССТФ и регуляторных районов) состоит в возможности их исследования в масштабе генома генов с использованием современных технологий массового параллельного секвенирования. Программный комплекс ICGenomics [44] качественно дополняет существующие методы анализа нуклеотидных последовательностей. Научная ценность работы связана с количественными оценками контекстной структуры геномных последовательностей в эмбриональных стволовых клетках, что позволяет уточнить молекулярные механизмы поддержания плюрипотентности и дифференцировки.
Программы и материалы, разработанные в ходе подготовки диссертации, доступны для научно-образовательных целей в Интернете на сайте ИЦиГ СО РАН по адресам: http://bioinformatics.bionet.nsc.ru/, http://www-bionet.sscc.ru/icgenomics/, http://wwwmgs.bionet.nsc.ru/mgs/programs/complexity/, http://pixie.bionet.nsc.ru/ratdna/rat/index.php, http://conf.nsc.ru/sbb2013.
Благодарности Автор выражает глубокую признательность научному консультанту академику РАН Колчанову Н.А., сотрудникам ИЦиГ СО РАН Д.А. Афонникову и В.А.
Иванисенко за помощь в подготовке работы и обсуждение научных результатов, В.А.
Кузнецову за научную дискуссию на ранних этапах работы. Автор благодарен зарубежным коллегам Guoliang Li, Yijun Ruan, Ed Liu, Neil Clarke, Bing Lim, Huck-Hui Ng за позитивный опыт работы и научного общения в международном научном коллективе.
Список сокращений БД – база данных ИПСК – индуцированные плюрипотентные стволовые клетки Кб – килобаза, тысяча пар нуклеотидов Мб – мегабаза, миллион пар нуклеотидов нт – нуклеотид НТП – нетранслируемая последовательность п.о. – пара оснований ДНК ССТФ – сайты связывания транскрипционных факторов т.п.н. – тысяча пар нуклеотидов ТФ – транскрипционный фактор ЭОПК – экспериментальный образец программного комплекса ЭСК – эмбриональные стволовые клетки Принятые англоязычные термины 3C (Chromosome Conformation Capture) – определение структуры хромосом ChIA-PET (Chromatin Interaction Analysis by Paired-End-Tag sequencing) – метод анализа взаимодействий хроматина с помощью секвенирования парных концов ChIP (Chromatin ImmunoPrecipitation) – иммунопреципитация хроматина ChIP-chip – технология иммунопреципитации хроматина на микрочипе ChIP-PET (Chromatin ImmunoPrecipitation - Paired-End-Tags) – технология иммунопреципитации хроматина c использованием парных концов ДНК ChIP-seq – технология иммунопреципитации хроматина с последующим секвенированием FISH – флюоресцентная гибридизация in situ H3K14ac – модификация гистонов – ацетилированный лизин 14 гистона Н H3K27me3 – метилированный лизин 27 гистона Н H3K4me3 – метилированный лизин 4 гистона Н H3K9ac – ацетилированный лизин 9 гистона Н Hi-C – метод определения конформаций хромосом в ядре клетки HMM (Hidden Markov models) – скрытые марковские модели NGS (Next Generation Sequencing) – высокопроизводительное геномное секвенирование (секвенирование следующего поколения) Введение В данной Главе представлен обзор литературы по современным направлениям исследований компьютерной геномики, технологиям экспериментального определения сайтов связывания транскрипционных факторов в геноме, методам анализа регуляции экспрессии генов эукариот, и соответствующим алгоритмам биоинформатики и базам данных. Глава содержит разделы, посвященные общим задачам компьютерного анализа геномных данных и проблемам компьютерного анализа данных геномного секвенирования. Описаны продолжающиеся международные проекты геномных исследований, направленные на создание аннотации функциональных элементов генома человека и основных модельных объектов - «1000 геномов», ENCODE [8] и modENCODE [65, 66], и доступные в Интернете базы данных геномной информации, включающие исходные данные секвенирования - GEO NCBI [34], GenBank [67], Ensembl [35]. Кратко представлены основные биоинформационные алгоритмы поиска гомологии, реконструкции филогенетических деревьев, статистические методы и алгоритмы предсказания сайтов связывания и регуляторных элементов в нуклеотидных последовательностях, необходимые для дальнейшего исследования.
Отдельный раздел Главы посвящен описанию молекулярных механизмов транскрипции эукариот, организации комплекса РНК-полимеразы II, регуляции транскрипции посредством белковых транскрипционных факторов. Представлена классификация регуляторных районов генов - промоторов и энхансеров, иерархическая организация регуляторных районов транскрипции генов эукариот.
В следующем разделе Главы показаны современные методы измерения экспрессии генов на уровне транскрипции: экспрессионные микрочипы (микроэрреи), секвенирование транскриптом (RNA-seq). Представлены компьютерные методы исследования регуляторных районов и сайтов связывания транскрипционных факторов, распознавания сайтов в нуклеотидных последовательностях по обучающим выборкам.
Описаны базовые экспериментальные технологии и полногеномные методы определения сайтов связывания транскрипционных факторов на основе иммунопреципитации хроматина - ChIP-seq и ChIP-PET [11, 15, 17, 18].
Дано описание групп транскрипционных факторов, исследование которых важно для медицинских приложений - онкогенов при раке и факторов поддержания плюрипотентности в эмбриональных стволовых клетках. Описаны проблемы исследования генов и поиска генов-мишеней действия кодируемых ими белков для транскрипционных факторов p53, STAT1, MYC [68], рецептора эстрогенов ER. В связи с исследуемыми задачами освещена роль регуляции транскрипции этих генов в транскрипционных факторов плюрипотентности в эмбриональных стволовые клетках репрограммирования соматических клеток, представлены имеющиеся данные по факторам OCT4, NANOG, SOX2 и ряду других.
В конце Главы представлен обзор имеющихся данных по проблеме исследования контактирующих участков хромосом в ядре клетке и регуляции транскрипции.
Описаны методы определения хромосомных контактов с помощью микроскопии и флюоресцентной in situ гибридизации (FISH), а также методы определения хромосомных контактов с помощью секвенирования: 3С, Hi-C и ChIA-PET.
В заключении Главы сформулированы возникающие задачи компьютерного секвенирования и иммунопреципитации хроматина.
1.1. ЗАДАЧИ КОМПЬЮТЕРНОГО АНАЛИЗА ГЕНОМНЫХ
ДАННЫХ
1.1.1. Международные проекты геномных исследований Полногеномное картирование и функциональная аннотация регуляторных последовательностей генов эукариот имеют большое значение для молекулярной биологии [28]. В целом, проблема компьютерного определения функции регуляторных районов по последовательности ДНК связана с неоднозначностью кодирования генетической информации [26, 31]. Участки ДНК, с которыми связываются транскрипционные факторы, не описываются нуклеотидной последовательностью однозначно. Сайты связывания РНК-полимераз, участки начала трансляции, регуляторные районы транскрипции генов, имеют еще более сложное строение, часто состоят из нескольких блоков, находящихся на варьирующих расстояниях.функциональных сигналов - сайтов связывания ТФ и регуляторных районов - требует применения более совершенных методов биоинформатики - методов распознавания образов, статистических подходов и вычислительных алгоритмов, связанных с обработкой огромных массивов информации. Прогресс в этой области зависит от уровня накопленных молекулярно-биологических знаний, экспериментальных и компьютерных методик, и смещается в настоящее время от предсказательных компьютерных моделей в сторону полногеномного анализа экспериментальных микроскопии, протеомики [28, 69]. Становится все более важен статистический анализ интегрированных компьютерных моделей [8, 13], а не только компьютерное предсказание на основе отдельных статистических характеристик, таких как физикохимические свойства ДНК, или частоты олигонуклеотидов.
Стремительно растут темпы исследований по секвенированию геномной ДНК [5, 67] (рис. 1.2). В настоящее время доступны последовательности более 24 тысяч полностью секвенированных геномов микроорганизмов и полутора тысячах геномов эукариот (http://www.ncbi.nlm.nih.gov/genome/browse/).
Число последовательностей Рис. 1.2. Рост объема геномных данных в мире по информации Европейского Института Биоинформатики (начало 2014 г.), накопленной в архиве секвенированных нуклеотидных последовательностей SRA (Sequence Read Archive) (http://www.ebi.ac.uk/ena/about/statistics).
По оси Y слева - число нуклеотидных последовательностей (серый цвет) в банке данных SRA (Sequence Read Archive), справа - число нуклеотидов (черный цвет), в логарифмической шкале. Общее число нуклеотидных последовательностей в банке данных - 4.6 триллиона, число нуклеотидов - 583 триллиона.
Проект «1000 геномов» уже содержит данные о более чем тысяче индивидуальных (персональных) геномов человека. Полностью секвенированы эукариотические геномы ряда растений, животных, включая геном мыши Mus musculus, геномы дрожжей S. cerevisiae и S. pombe, некоторых видов рыб, включая Danio rerio.
Продолжается исследование геномов множества других видов, имеющих большое сельскохозяйственное или медицинское значение. Растет как общий объем геномных последовательностей, так и число видов, последовательности геномной ДНК которых (http://www.ncbi.nlm.nih.gov/bioproject/). Общий объем данных по нуклеотидным последовательностям удваивается каждые 2-3 года (см. рис. 1.2), а новые полностью секвенированные последовательности бактериальных геномов становятся доступными в среднем раз в две недели.
Развитие сети Интернет, появление общедоступных молекулярно-генетических баз данных большого объема предоставляет новые возможности и ставит новые задачи, связанные с исследованием полных геномов, поиском регуляторных районов транскрипции генов и их аннотации [7].
высокопроизводительного секвенирования [2]: параллельное пиросеквенирование на (http://www.illumina.com), использующая оптическое сканирование флюоресценции меченых нуклеотидов в клонированных колониях молекул ДНК на твердой поверхности, и технология секвенирования с помощью лигирования ABI (Applied Biosystems) (http://www.appliedbiosystems.com) SOLiD (Sequencing by Oligonucleotide Ligation). Перспективны новые технологии Ion Torrent, использующие детекцию ионов водорода во время полимеризации ДНК на гиперчувствительном сенсоре [70]. Эта компания поглощена компанией Life Technologies, которая в свою очередь поглощена компанией ThermoFisher (www.thermofisher.com). Соревнование производителей оборудования секвенирования ДНК, гонка технологий, свидетельствует о большой практической значимости полногеномных методов исследования, актуальности геномных исследований в целом.
Секвенирование ДНК на наносферах (nanoball sequencing) компании Complete Genomics (www.completegenomics.com) основано на циклической амплификации фрагментов геномной ДНК по принципу «катящегося кольца». В 2013 году компания Complete Genomics поглощена Пекинским Институтом Биоинформатики (BGI) крупнейшим международным центром секвенирования. Компания Pacific Biosciences (PacBio) предлагает альтернативную технологию определения последовательности одиночной молекулы ДНК (технология SMRT) при считывании ДНК-полимеразой (www.pacificbiosciences.com/). Каждая из технологий имеет свои стандарты представления данных, что требует новых компьютерных решений.
Общий тренд в технологиях секвенирования вне зависимости от физических принципов определения нуклеотидной последовательности состоит в обратной связи объемов и стоимости секвенирования, длины полученных последовательностей ДНК и требованиям к биоинформационной составляющей анализа: чем ниже цена за секвенирования за нуклеотид (за мегабазу), тем выше производительность технологии секвенирования. Чем выше производительность, тем короче получающиеся фрагменты ДНК. Чем короче прочтения ДНК (секвенированные последовательности), тем сложнее последовательностей (контигов), сложнее математический аппарат и выше требования к компьютерным вычислениям. Так, секвенирование по технологии Roche (www.454.com/) позволяет получать последовательности до 300 нуклеотидов, в то время как следующие технологии, такие как SOLiD, – не более 70-100 нуклеотидов и даже 35-50 нт в первых моделях. Использование более коротких последовательностей ДНК ставит технически более сложные задачи биоинформационного анализа, выравнивания, ассемблирования и картирования последовательностей.
Представление геномных последовательностей в базах данных Гены эукариот имеют сложную «мозаичную» экзон-интронную организацию [27], включают регуляторные районы, что требует изучения с помощью современных полногеномных методов [71] и баз данных [72].
В целом, геномы эукариот характеризуются низкой плотностью кодирующих районов [73], так, для человека она составляет менее 2%. В интронах и межгенных спейсерах располагаются различные типы повторяющихся последовательностей [74], информация о которых представлена в специализированных базах данных, таких как RepeatMasker [75-77].
1.1.2. Статистические методы и алгоритмы Классическими алгоритмами биоинформатики являются алгоритмы поиска совпадений в нуклеотидных последовательностях и выравнивания. Выравнивание последовательностей – это процедура сравнения двух (парное) или более (множественное выравнивание) последовательностей путем поиска серий (блоков) символов, находящихся в последовательностях в том же порядке [27, 78], с помощью записи их в две строки с пробелами. Метод поиска выравнивания с учетом вставок и делеций впервые был предложен в работе Нидльмана и Вунша [79]. Определяется функция сходства F, которая учитывает число гомологичных совпадений, а также замены и вставки. В качестве параметров метода вводятся веса, увеличивающие функцию F при обнаружении гомологичного совпадения и штрафы за замены и вставки (делеции), уменьшающие F:
где Km, Kd, Kc – количество совпадений (m), делеций (d) и замен (c), Vm, Vd, Vc – параметры, характеризующие веса совпадений, делеций и замен, соответственно.
Для двух сравниваемых последовательностей строится точечная матрица гомологии. Задача решается с помощью динамического программирования. Для построения оптимального пути, соответствующего максимуму функции сходства, восстанавливается весь путь в точечной матрице гомологии, соответствующий выравниванию:
Был разработан ряд эвристических методов, имеющих в своей основе идеи метода выравнивания Нидльмана-Вунша [80], и широко применяющихся для массового последовательностей разработано несколько алгоритмических компьютерных приемов.
Метод быстрого поиска повторов в тексте, предложенный в работе [81], получил название метода l-граммного разложения. Такое технологическое решение применяется как для быстрого поиска гомологий в банках данных, так и для поиска повторов в протяженных последовательностях [82, 83]. Другой подход, используемый для быстрого поиска гомологий – суффиксные деревья. Этот метод может использоваться как для быстрого поиска гомологий, так и для выявления консервативных мотивов в выборках функциональных последовательностей [84].
Поиск гомологий на основе алгоритмов выравнивания FASTA и BLAST Наиболее распространенными для массового анализа являются программы FASTA и BLAST. Программа FASTA (FAST Alignment and Search Tool - all), основанная на динамическом поиске совпадений, была разработана для быстрого поиска гомологий между двумя белковыми или нуклеотидными последовательностями [85]. Полностью совпадающие (инициирующие) участки расширяются с учетом возможных несовпадений, вставок и делеций. Далее алгоритм выполняет выравнивание картирования коротких последовательностей высокопроизводительного секвенирования (http://drfast.sourceforge.net/; http://mrsfast.sourceforge.net/) Алгоритм BLAST (Basic Local Alignment Search Tool) работает на порядок быстрее FASTA [78]. В основе метода лежит понятие пары сегментов с высоким счетом (HSP – high-scoring segment pair), т.е. такие участков одинаковой длины, для которых получено значение функции сходства больше некоторого порогового значения.
Программа BLAST (http://blast.ncbi.nlm.nih.gov/) является мировым стандартом, алгоритм имеет огромное число цитирований [69]. Программа может быть инсталлирована локально и настроена на заданные пользователем геномные базы данных. В BLAST для поиска гомологии в базах белковых последовательностей могут использоваться алгоритмы BLASTP (парное выравнивание последовательностей), PSIBLAST (позиционно специфический итерационный BLAST), PHI-BLAST (поиск паттернов, инициируемый BLAST). В PSI-BLAST для выравнивания используются позиционно специфические матрицы весов (PSSM).
Кроме стандартных алгоритмов разработан ряд оптимизированных программ быстрого поиска протяженных последовательностей, такие как BLAT (BLAST-like alignment tool) (http://genome.ucsc.edu/cgi-bin/hgBlat) [86], картирования коротких последовательностей, реализации алгоритмов поиска для параллельной компьютерной архитектуры [2, 87].
Реконструкция деревьев сходства Набор выровненных нуклеотидных или аминокислотных последовательностей может быть использован для восстановления (построения) филогенетического дерева.
Филогенетическое дерево – это бинарный (древовидный) граф, отражающий гипотетическую картину дивергенции последовательностей. Среди методов построения деревьев можно выделить группу матричных методов [88], метод объединения соседей [89], метод максимальной экономии, метод максимального правдоподобия и некоторые другие [27, 90].
Существуют сотни пакетов программ, предназначенных для выполнения той или иной части филогенетического анализа (на 2013 год было представлено около программ на http://evolution.genetics.washington.edu/phylip/software.html). Отметим, в частности, пакеты программ филогенетического анализа MEGA [91], PAML [92] и пакет VOSTORG, разработанный в ИЦиГ СО РАН [93, 94]. Распространен пакет программ PHYLIP [95], использующийся для анализа матриц расстояний между последовательностями и построения филогенетических деревьев. Разработаны конвейеры программ для анализа эволюционных расстояний, такие как SAMEM (http://pixie.bionet.nsc.ru/samem/) [96].
Сравнение точности методов распознавания Для сравнения методов предсказания (распознавания) функциональных элементов ДНК используются меры точности распознавания, пришедшие из математических дисциплин [97]. В терминологии теории распознавания образов функциональные районы (промоторы, сайты связывания и т.д.) соответствуют классу «Да», участки ДНК, не выполняющие такой функции, – классу «Нет». Таблица сопряженности результатов предсказания размера 2 2 содержит стандартную классификацию и терминологию, используемую при сравнении точности предсказания.
(Класс «Да») (Класс «Нет») Значения из таблицы сопряженности используются для подсчетов величин, характеризующих статистическую значимость (величина р) получения наблюдаемого результата. Для оценки точности предсказания используют ошибки первого и второго рода. Ошибка первого рода E1 – недопредсказание, доля ложно предсказанных объектов класса «Да», E1=FP/(FP+TP). Ошибка второго рода – перепредсказание, доля ложно предсказанных объектов класса «Нет» E2=FN/(TN+FN). Статистическая значимость может быть оценена по точному критерию Фишера [98].
Другие меры оценки точности методов предсказания – чувствительность Sn и специфичность Sp [88]. Чувствительность – доля правильных предсказаний среди всех реальных объектов, Sn=TP/(TP+FN), а специфичность – доля правильных предсказаний по отношению ко всем полученным предсказаниям Sp=TP/(TP+FP). Специфичность – обратная величина к ошибке первого рода, Sp=1-E1.
классификаций TPR (True Positive Rate). Специфичность Sp классификации также называют долей ложных положительных классификаций FPR (False Positive Rate).
Для сравнения методов распознавания используют как ошибки первого и второго рода, так и чувствительность и специфичность. Для сравнения точности методов по корреляционный коэффициент ошибок:
Площадь под кривой ошибок Стандартом оценки точности распознавания/предсказания в биоинформатике при варьировании порога является площадь под кривой ошибок (ROC-AUC), которая принимает значения в интервале [51, 99]. Кривая ошибок или ROC-кривая – график, позволяющий оценить качество бинарной классификации, отображает зависимость доли верных положительных классификаций (TPR) от доли ложных положительных классификаций (FPR) при варьировании порога решающего правила.
Термин ROC (Receiver Operating Characteristic) - операционная характеристика приёмника пришёл из теории обработки сигналов. ROC-кривая показывает зависимость TPR от FPR при варьировании порога распознавания (Рис. 1.3).
чувствительность транскрипционных факторов MYC и p53 (TP53) в геноме человека [99].
Кривая проходит из точки (0,0), соответствующей максимальному значению порога (все объекты классифицируются как отрицательные, и ошибки возникают на всех положительных объектах, FPR=0, TPR=0), в точку (1,1), соответствующую минимальному значению порога (все объекты классифицируются как положительные, FPR=1, TPR=1). Случайное распознавание соответствует прямой линии, площадь под кривой равна 0.5. Лучший вариант — это кривая, проходящая на графике через точки (0,0); (0,1); (1,1).
На рисунке 1.3 приведен пример кривой ошибок для предсказания сайтов связывания транскрипционных факторов в геноме человека [99]. Видно, что кривая ошибок выше для сайтов связывания MYC (левая панель), чем для сайтов связывания p53 (правая панель), и, соответственно распознавание сайтов MYC точнее [99].
Площадь под ROC-кривой AUC (Area Under Curve) является агрегированной характеристикой качества классификации, не зависящей от соотношения ошибок. Чем больше значение AUC, тем «лучше» модель. Данный показатель используется для сравнительного анализа нескольких моделей классификации. Используется также бутстреп (bootstrap) анализ, когда составляется обучающий набор из случайно выбранных элементов выборки и проверка предсказания на оставшихся данных. Такой анализ будет представлен в Главе 4 данной работы.
Для распознавания и классификации нуклеотидных последовательностей на основе набора характеристик (контекстных, физико-химических или полногеномных) используются регрессионные модели, в том числе логистическая регрессия.
Обобщённые линейные модели, называемые также обобщёнными аддитивными моделями, можно рассматривать как обобщение криволинейной регрессии.
Логистическая регрессия — частный случай обобщённой линейной модели, если взять логит-функцию связи P=1/(1+e-y), где y является линейной комбинацией независимых переменных y=w1x1+w2x2+...+w0. С помощью анализа зависимой переменной, принимающей значения от 0 до 1 (имеющей смысл вероятности) можно оценить ошибки классификации. Логистическая регрессия применяется для решения задач классификации и позволяет оценивать вероятности принадлежности объекта рассматриваемым классам.
Вычислительные процедуры в масштабе генома Отметим, что оценка параметров сложных распределений, симуляция предсказаний для случайных последовательностей в геномах требует сложных вычислительных процедур. Так при генерации псевдослучайных чисел, соответствующих например позициям в нуклеотидной последовательности генома, необходим датчик чисел, избегающий повторов и периодичностей, такой как «Mersenne Twister» [100]. Виртуальное пространство для генерации распределения позиций сайтов в геноме человека (соответствующее линейным) позициям на хромосомах, составляет около 3 гигабаз - значительно больше, чем может произвести датчик случайных чисел без повторов. Датчик случайных чисел на компиляторе UNIX C++ может дать периодичность в многократно повторяемых симуляциях (поскольку рассчитан на генерацию до 231=~2Гб, что меньше моделируемого пространства). Соответствие качества работы генерации случайных чисел без повторов этого датчика для моделирования биологических систем было показано в работе [101].
Статистический и комбинаторный анализ нуклеотидных последовательностей Изучение нуклеотидных слов - коротких последовательностей, позволяет анализировать регуляторные последовательности генов, выдвигать гипотезы о функциональной роли отдельных фрагментов генетического текста [71, 102], в том числе регуляторных районах генов. Исторически одним из первых представление о генетических языках, содержащих нуклеотидные слова, было введено В.А. Ратнером в 1970-х [103]. Плодотворным оказался подход, связанный с лингвистическими представлениями текста и математической теорией кодирования информации [104В связи с исследованиями структуры генетических текстов Э.Н. Трифоновым разрабатывалась теория множественности кодов, содержащихся в генетических текстах [104, 108]. В первичной структуре белка представлена информация о его пространственной структуре и локализации функциональных сайтов. В первичной структуре мРНК, помимо информации о кодируемой аминокислотной последовательности, присутствует информация о вторичной структуре [109, 110]. На уровне гена, кодирующего эту мРНК, есть информация о локальной конформации ДНК в виде взаимного расположения пуриновых и пиримидиновых пар, а также информация о локализации нуклеосом – в виде участков специфического связывания с гистонами [104, 111, 112]. Таким образом, в пределах природного генетического текста может быть записано несколько генетических сообщений, определяющих различные аспекты структурно-функциональной организации макромолекул [110, 112]. Одновременная запись возможна лишь в случае, если эти генетические сообщения совместимы [113].
Так, триплетный код допускает наложение нескольких слабо позиционированных структурных сигналов в последовательности путем синонимичных замен [110, 112].
Важной универсальной характеристикой геномных последовательностей, является сложность текста [114, 115]. Интерес представляет оценка сложности генерации (порождения) текста в виде минимального числа операций копирования, необходимых для воспроизведения последовательности по ней самой (по методу Лемпеля и Зива). Такие операционные меры, адаптированные к последовательностям ДНК, были предложены В.Д. Гусевым и соавторами [115, 116], и развиты в работе автора диссертации [114]. Сложность текста может быть определена различными способами, основанными на алгоритмических оценках [105, 106], оценках энтропии Шеннона [117], разнообразии словаря различных слов длины k (k-мер) [118]. Поиск участков низкой сложности в нуклеотидных последовательностях связан не только с теоретическими оценками распространенности повторов в геноме, но и с задачами анализа результатов высокопроизводительного геномного секвенирования, оценок уникальности картирования коротких последовательностей в геноме [57].
Разработанные автором компьютерные программы оценки сложности текста [114] применялись в данной работе для фильтрации данных ChIP-seq, анализа ошибок в прочтениях ДНК при секвенировании.
1.2 ТРАНСКРИПЦИЯ ГЕНОВ ЭУКАРИОТ
1.2.1. Транскрипция и транскрипционные факторы Первая стадия считывания генетической информации - транскрипция ДНК проходит с участием РНК-полимераз и зависит от других белков – факторов транскрипции [73]. Синтез РНК, не зависящий от присутствия регуляторных молекул, называют базальной транскрипцией. Полногеномные методы исследования [119, 120] показали, что связанные с проксимальными промоторами РНК-полимеразы находятся одновременно примерно на 30% генов в геноме человека.Регуляция работы генов в клетках эукариот координируется с помощью белковых факторов в зависимости от типа ткани, стадии развития организма, фазы клеточного цикла [73, 121]. Экспрессия генов эукариот связана с особенностями нуклеосомной упаковки хроматина, метилированием ДНК, интенсивностью сплайсинга, полиаденилирования, стабильностью мРНК в цитоплазме, посттрансляционными модификациями, внутриклеточным транспортом и скоростью деградации белка [122Ключевая роль в регуляции экспрессии генов принадлежит транскрипции, запускающей цепочку молекулярных процессов [126, 127]. Ядерные белковые факторы транскрипции способны связываться с последовательностями ДНК, оказывая влияние на эффективность экспрессии генов, расположенных в разных участках генома [128]. В современных исследованиях встают задачи определения сайтов связывания транскрипционных факторов в масштабе генома, определения на этой основе геновмишеней и реконструкции регуляторных генных сетей.
ДНК-белковые контакты включают водородные связи и Ван-дер-Ваальсовы расположенными на поверхности белка, и атомами азотистых оснований сайта ДНК Разработана структурная классификация ДНК-связывающих доменов, [128].
содержащая классы основных факторов (в том числе «лейциновая застежка»), класс координированных цинком ДНК-связывающих доменов, класс «спираль-поворотспираль» (включая гомеодомен) и класс бета-укладки с контактами по малой бороздке ДНК [129]. Большинство транскрипционных факторов контактируют со своим сайтом связывания по большой бороздке двойной спирали ДНК.
Основная (базальная) транскрипционная машина включает в себя РНКполимеразу II и белковые комплексы - основные факторы транскрипции (GTF - от английского General Transcription Factors) TFIIA, TFIIB, TFIID, TFIIE, TFIIF, TFIIH и TFIIK (рис. 1.5). Сборка инициирующего комплекса начинается со связывания фактора транскрипции TFIID с ТАТА-боксом. Сначала с ТАТА-боксом связывается одна из субъединиц TFIID - TATA-связывающий белок (или ТВР - TATA-box binding protein) [130-132]. Белки - модуляторы транскрипции могут взаимодействовать с другими общими транскрипционными факторами [133, 134].
Рис. 1.4. Схема активирующих взаимодействий между активаторами, ко-активаторами и медиатором [135].
Медиатор транскрипции - это комплекс белков, связывающийся с С-концевым доменом (CTD) РНК-полимеразы II и образующий с ним полный фермент холофермент (holoenzyme) [136]. Медиатор необходим для связи между РНКполимеразой II и белками-активаторами транскрипции [137]. Существует ряд белков регуляторов транскрипции, или ко-факторов, которые не контактируют непосредственно с ДНК, а взаимодействуют с другими факторами, связанными с ДНК, и могут быть либо ко-активаторами, либо ко-репрессорами.
Исследование и описание взаимодействий ко-активаторов с ДНК в регуляторных районах генов в масштабе генома представляет собой важную фундаментальную научную проблему, для решения которой необходим компьютерный анализ полногеномных экспериментальных данных.
Комплекс РНК-полимеразы II Существует три типа эукариотических РНК-полимераз: I, II и III. РНК-полимераза II транскрибирует белок-кодирующие гены [138]. При выходе из клеточного ядра молекулы мРНК, транскрибированные полимеразой II, проходят серию ковалентных модификаций, определяющих их функциональную специализацию и отличающих их от транскриптов, синтезированных другими РНК-полимеразами.
ТАТА-связывающий белок (TBP) – ключевой элемент механизма инициации транскрипции эукариот, входящий в состав комплекса TFIID, также необходим для осуществления транскрипции РНК-полимеразами I и III [126]. Комплекс TFIID - общий фактор транскрипции, состоящий из нескольких субъединиц, который связываясь с промотором обеспечивает формирование инициаторного комплекса [139, 140]. В состав TFIID входят до 12 факторов ТАФ (TBP-ассоциированные факторы, или TAF - TBPassociated factors).
Исследование комплекса РНК-полимеразы II, анализ распределения такого связывания в геноме важны для исследования промоторных районов белоккодирующих генов.
1.2.2. Методы измерения экспрессии генов Большое значение для полногеномного анализа имеет интеграция полногеномных данных по экспрессии генов (транскриптомные данные) с данными по расположению генов на хромосомах, их характеристикам, паттернам экспрессии в тканях организма [141]. Так, анализ групп высокоэкспрессирующихся генов показал, что они имеют Количественные оценки экспрессии генов эукариот должны опираться на современные полногеномные методы.
транскрибированной мРНК) может быть выполнено с помощью ПЦР в реальном времени, с помощью экспрессионных микрочипов [144], с помощью технологий EST (аббревиатура от Expressed Sequence Tags), SAGE (Serial Analysis of Gene Expression) [145, 146]. За последние годы было предложено несколько технологий анализа экспрессии генов как с помощью экзонных микрочипов, так и с помощью тотального секвенирования (RNA-seq) [4]. Разработаны карты транскриптом для различных тканей человека [141, 147].
Микрочипы получили большое распространение несколько лет назад, но в настоящее время технология микрочипов отходит, уступая по эффективности технологиям секвенирования RNA-seq. Тем не менее, накопленный за последние годы значительный массив экспериментальных, прежде всего клинических, данных об экспрессии генов на микрочипах, делает необходимым разработку оптимальных компьютерных методов для использования таких данных.
ДНК-микрочип (микропластина, или микроэррэй, от англ. - microarray) — это комплексная технология, используемая в молекулярной биологии и медицине.
Микрочип состоит из нескольких (от десятков до тысяч) микроскопических ячеек на пластинке (чипе) содержащих дезокси-олигонуклеотиды. Каждая ячейка содержит ДНК специфической последовательности, которая используется для гибридизации с кДНК или мРНК. Эксперимент проводится во многих ячейках одновременно для заданного множества транскрибирующихся последовательностей (проб). Гибридизация зонда и мишени регистрируется и количественно определяется при помощи флюоресценции или хемилюминесценции.
Микрочипы отличаются по конструкции, особенностям работы, эффективности, технологическим подходам. Обычно, в микрочипе зонды ковалентно прикрепляются к твердой поверхности — стеклянному или кремниевому чипу. Распространены микрочипы компаний Affymetrix (www.affymetrix.com/), Illumina (www.illumina.com), Agilent (http://www.home.agilent.com/), NimbleGen (www.nimblegen.com/), CodeLink (www.appliedmicroarrays.com).
компанией Illumina, используют микроскопические шарики вместо твердых поверхностей. Отметим различие технологических платформ: одноцветовой микрочип (one-color) компании Affymetrix [148] и двухцветовые чипы (two-color) компаний NimbleGen и Agilent. Методы измерения уровней экспрессии генов на основе таких микрочипов получили широкое распространение в медицинских исследованиях [149Отмечена низкая корреляция между измеренной экспрессией одних и тех же генов на микрочипах и с помощью других технологий [146].
Основной статистической задачей обработки данных экспериментов на микрочипах является определение дифференциально экспрессирующихся генов.
Разработан ряд пакетов для решения этой задачи, таких как SAM (Statistical Analysis of Microarrays) [153]. Не менее важны и задачи процессинга данных, адекватного определения сигнала проб на микрочипе, рассмотренные в настоящей работе.
Распространенные коммерческие платформы микрочипов (в частности Affymetrix) имеют ряд технических недостатков, связанных с несоответствием проб и генов, для измерения транскрипции которых предназначены эти пробы [46].
Технология синтеза коротких олигонуклеотидных зондов (25 п.н.) непосредственно на поверхности микрочипа in situ с использованием литографических масок была изготовления микрочипов GeneChip. Исходно до 2003 г. был разработан микрочип GeneChip U133A, дополненный позднее чипами U133B и U133 plus 2, более полно соответствующими всем известным и проаннотированным на тот момент генам в геноме человека.
Олигонуклеотидная матрица GeneChip использует наборы синтезированных in situ олигонуклеотидных проб, по 11–20 проб в наборе, каждая размером нуклеотидов, для представления транскриптов генов или их изоформ. Для каждого исследуемого гена использованы фрагменты-представители (initial target sequences) длиной 150–450 п.н. для выбора и локализации олигонуклеотидных проб. Уровень экспрессии гена определяется суммой данных всего набора проб (probeset) [154].
Сигнал от пробы с совершенным совпадением всех нуклеотидов учитывается после вычитания неспецифического сигнала кросс-гибридизации от пробы с одним http://www.affymetrix.com/support/).
Проблема анализа транскрипции с помощью этого микрочипа в целом связана с рядом технических ограничений и ошибок при создании технологии. Дизайн проб (исходный выбор производителем микрочипов локализации в гене и структуры олигонуклеотидных проб) может не соответствовать целевому транскрипту (гену) и содержать ряд технических проблем, связанных как с гибридизацией, так и с аннотацией – неверное указание гена-мишени, неоднозначность соответствия один набор проб–один ген. Такой дизайн олигонуклеотидных проб может влиять на регистрацию сигналов гибридизации, нормализацию данных, снижать воспроизводимость экспериментов, вести к противоречивым результатам анализа одних и тех же данных [151, 156-159].
Ранее была выполнена независимая аннотация наборов проб микрочипов Affymetrix на основе картирования нуклеотидных последовательностей проб на референсные последовательности генома человека [150, 151, 156]. Выявлен ряд несоответствий в аннотации наборов проб для идентификации генов; такие несоответствия могут затрагивать до 30–50 % наборов проб [151, 157, 159].
Соревнование в сфере технологий производства микрочипов, технологий измерения сигналов экспрессии генов дало большой толчок научным исследованиям и огромный фактический материал. Отметим еще раз, что за последние годы на смену микрочипам приходят все более совершенные технологии полного секвенирования транскриптом, имеющие ряд принципиальных научных преимуществ, в частности, по способности определения новых вариантов транскриптов гена, по динамической шкале измерения уровня транскрипции [4, 14]. Тем не менее, микрочиповая технология позволяет достаточно надежно и относительно недорого определять дифференциально экспрессирующиеся гены за счет репликации экспериментов [14], и требует разработки специализированных компьютерных инструментов.
В связи с представленными проблемами измерения экспрессии генов встает задача получения статистических оценок качества наборов проб микрочипов, в частности платформы Affymetrix U133 Plus 2.0 для генов человека. Компьютерная оценка уникальности нуклеотидных последовательностей проб микрочипа и соответствия аннотации генов в геноме должна быть подкреплена анализом экспрессии систематизированы и представлены в общедоступной компьютерной базе данных вместе со статистической оценкой величины и качества измеряемых сигналов на микрочипе, что и было выполнено в настоящей работе.
перекрывающихся с цис-антисенс транскриптами генов человека и описание таких транскриптов в целом. Феномен цис-антисенс транскрипции в геноме человека должен быть подкреплен микрочиповыми данными. Сходство нуклеотидных проб c транскриптами повторяющихся последовательностей SINE и LINE в геноме позволяет оценить как качество проб микрочипа, так и возможную экспрессию транспозонов, детектируемую на микрочипе в опухолевых клетках. По методическим и техническим причинам геномные повторы обычно исключаются из дизайна микрочипов [160], в частности из-за избыточности мобильных элементов в геноме и сложности подбора уникальных проб. Таким образом, их потенциальная транскрипционная активность остается недостаточно охарактеризованной, несмотря на многочисленные наблюдения присутствия транскрипции в различных тканях при заболеваниях человека [161], в том числе при раке молочной железы [162].
1.3 РЕГУЛЯТОРНЫЕ УЧАСТКИ ГЕНОВ: ПРОМОТОРЫ И
ЭНХАНСЕРЫ
1.3.1. Промоторы и энхансеры Промотором называют последовательность ДНК, связывающую РНК-полимеразу и служащую отправной точкой транскрипции [163]. В целом, у многоклеточных эукариот, в пределах 100-200 п.н. перед стартом транскрипции выявлена сложная схема последовательностями - мотивами или боксами [164]. Минимальный промотор (или «коровый» промотор, от англ. core) содержит ряд коротких функционально значимых последовательностей размером 5-25 п.о. [126, 165]. В коровом промоторе наиболее полно изучены ТАТА-бокс, инициатор (Inr-элемент), CAAT-бокс и GC-бокс [166] (рис.1.5). ТАТА-бокс представляет собой А/Т-богатую последовательность (TATAWAW) [167], расположенную на расстоянии 28-34 п.о. выше старта транскрипции. Inr-элемент содержит старт транскрипции. Выделяют также полипиримидиновый инициатор (TCT), BRE элемент (TFII-B Recognition Element), MTE элемент (Motif Ten Element), DPE элемент (Downstream Promoter Element) [168] и E-бокс. Отметим, что в промоторах эукариот в целом нет однозначной записи контекстных регуляторных сигналов и нет заранее заданной локализации этих сигналов [169], что ставит целую серию задач компьютерного поиска и распознавания таких регуляторных участков в геноме.
промоторные элементы
GCCCAATCT TATAAA
GGGCGG
Рис. 1.5. Структура промотора гена эукариот и основные промоторные элементы - GCбокс, CAAT-бокс, TATA-бокс, Inr. Адаптировано из [170].Традиционно, по наличию или отсутствию ТАТА-бокса промоторы делятся на две группы: ТАТА-содержащие и ТАТА-несодержащие [166, 171]. В отдельную группу выделяют промоторы, содержащие DPE элемент, являющийся функциональным аналогом ТАТА-бокса, который локализован в районе +30 относительно старта транскрипции [172].
Транскрипция генов, считываемых РНК-полимеразой III, определяется промотором, лежащим внутри гена. РНК-полимераза III способна к реинициации, транскрибирует гены в районах свободных от нуклеосом [173]. Промотор для РНКполимеразы I, транскрибирующей гены рибосомальных РНК, недостаточно охарактеризован, не удается составить для него общую схему регуляторных элементов [174].
Энхансер (от англ. enhancer) - это регуляторная последовательность нуклеотидов, усиливающая активность промоторов эукариот [163]. Энхансер значительно, в десятки раз, усиливает транскрипцию, причем это воздействие практически не зависит от расположения энхансера относительно контролируемого гена [175]. Энхансеры способны действовать на больших расстояниях (более нескольких тысяч п.н.), располагаясь как в 5'-, так и в 3'-конце районе, а также внутри гена в составе интронов [176, 177]. Энхансер гена альбумина находится перед промотором, у генов иммуноглобулинов регуляторные элементы расположены в интронах. Энхансер может быть расположен и ниже гена на большом расстоянии, как у бета-глобинового гена [178]. Транскрипция гена SHH человека [179] контролируется его энхансером, который расположен на расстоянии 1Mб и вложен в интронный район LmbR1. Точечная мутация в этом энхансере вызывает преаксиальную полидактилию, общее врожденное нарушение формирования конечностей у млекопитающих [180].
Потенциальные энхансеры могут быть определены экспериментально с помощью высокопроизводительных экспериментальных подходов [181][182], но нерешенной остается проблема сопоставления энхансеров и их генов-мишеней находящихся на удалении сотен килобаз. Многие дальние энхансеры могут быть вложены в интронные районы других дистально расположенных генов [183], делая неоднозначным соотнесение энхансеров их генам-мишеням.
В литературе обсуждаются два основных механизма действия энхансеров [163]:
сверхспирализованные хромосомные петли и непосредственные взаимодействия.
Полагают, что функциональные участки генома, содержащие один или несколько генов, образуют длинные петли, включающие десятки тысяч нуклеотидных пар ДНК.
Высказано предположение, что такие хромосомные петли закреплены в матриксе клеточного ядра и сверхспирализованы. В состав матрикса входит топоизомераза II, повидимому, определяющая топологию петли ДНК. Взаимодействие энхансера с белками может менять конформацию всей петли, включая удаленный от энхансера участок ДНК, в результате чего в составе петли изменяется локальная структура хроматина и облегчается транскрипция гена.
транскрипционных комплексов, с которых транскрипция инициируется и реинициируется. Такое предположение ставит проблему прямого экспериментального исследования контактирующих участков ДНК, взаимодействующих с комплексом полимеразы II, что было представлено в работе автора на основе технологии секвенирования ChIA-PET [12].
Ремоделирование нуклеосом также может быть одним из механизмов проявления энхансерной активности [163]. В целом можно сказать, что энхансер действует на любой ближайший к нему промотор. Показано, что проксимально расположенные друг к другу гены имеют тенденцию быть совместно регулированными независимо от функциональных различий между ними [184-186] (так называемые «нейтрально коэкспрессирующиеся кластеры»). Такие кластеры могут появляться в результате эффекта нейтральной коэволюции [187].
Факторы ремоделинга хроматина и белков, организующих структуру хроматина Ini1, Brg1, CTCF [188] и Rad21 [189] ассоциированы с энхансерными районами.
Показано, что Ini1 и Brg1, две субъединицы комплекса SWI/SNF, вовлечены в транскрипционные петли [190, 191].
Эффект регуляции достигается посредством сборки на последовательностях энхансера белкового комплекса, который иногда называют энхансеосомой [192], и его взаимодействия с основным транскрипционным комплексом путем белок-белковых взаимодействий [193]. Обсуждалась проблема стереоспецифичности во взаимодействии энхансерных белковых комплексов - то есть специфичность ориентации в цепи ДНК энхансера по отношению к промотору [175, 193].
Энхансеосома определяется как нуклеопротеиновый комплекс, состоящий из различных наборов сайтов связывания ТФ связанных напрямую или опосредованно с энхансерной ДНК [194, 195]. Прототипом энхансеосомы может служить вирусиндуцируемый энхансер гена интерферона- (IFN-). Этот энхансер связан субъединицами p50 и p65 NF-B, ATF-2, IRF-3, IRF-7, c-Jun, и архитектурным транскрипционным фактором HMGA. Атомная модель этого комплекса содержащего восемь этих факторов, связанных с ДНК была реконструирована на основе трех кристаллических структур [196].
Контрольные области генов (LCR) часто содержат множественные энхансерные модули, которые варьируют в размерах от 50 нуклеотидов до 1,5 Кб [197]. Каждый из этих модулей может активировать ген на определенной стадии развития или в определенном типе клеток. Один ген может содержать множество энхансерных модулей, каждый из которых вносит свой вклад в пространственную и временную регуляцию экспрессии гена. Энхансер в зависимости от белкового фактора может начать вести себя и как негативно действующий регуляторный элемент экспрессии гена - сайленсер (от англ. silencer).
В целом, трудно привести четкие различия между энхансерами и элементами промоторов эукариот. Так, в работе [198] на основе анализа большого набора тканей и клеточных линий человека показана возможность предсказания дистальных энхансеров в геноме на основе контекстных свойств промоторов (присутствия специфичных ССТФ). Регуляторные элементы генов, которые первоначально относили либо к промоторам, либо к энхансерам, обладают рядом общих функциональных характеристик, таких как присутствие сайтов связывания транскрипционных факторов, воздействие на экспрессию близлежащих генов. Возникает задача исследования энхансеров в масштабе генома, определения удаленных от генов регуляторных районов, в том числе с помощью полногеномных экспериментальных методов.
Иерархическая организация регуляторных районов эукариот Особенность регуляторных районов генов эукариот – их иерархическая организация. Два соседних ССТФ могут представлять композиционный элемент. В этом случае их совместное действие согласовано, то есть его комбинаторный эффект значительно отличается от действия каждого ССТФ в отдельности [199, 200].
5’-регуляторные районы генов эукариот характеризуются также большим размером, достигающим десятков тысяч п.o. [165], что на порядки больше максимального размера регуляторных районов прокариот.
Считывание с одного гена разных вариантов РНК называется альтернативной транскрипцией. Эта особенность регуляции лежит в основе механизма формирования большого разнообразия первичных транскриптов одного и того же генного локуса и, как следствие этого, разнообразия белков, кодируемых одним и тем же генным локусом. В настоящее время известны примеры первичных транскриптов, в которых сплайсинг может проходить по десяткам альтернативных путей [201, 202]. Так, у человека, более 42% генов имеют альтернативный сплайсинг пре-мРНК. Причем значительная их часть кодирует определенные типы молекул (например, клеточные рецепторы), а также белки, выполняющие системные функции в организме, в частности в иммунной и нервной системах [201].
Транскрипционная активность гена зависит от стадии клеточного цикла, функционального состояния клетки, ткани, органа, стадии индивидуального развития, транскрипционных факторов, которые взаимодействуют с регуляторными элементами конкретного гена. В результате формируется уникальный транскрипционный комплекс, обеспечивающий необходимый уровень транскрипции гена в конкретной клеточной ситуации [121, 123]. Блочно-иерархическая организация регуляторных районов генов эукариот (сайты, промоторы, дистальные регуляторные элементы) обеспечивает возможность гибкой регуляции транскрипции за счет включения/выключения отдельных элементов [124]. Примером сложной организации регуляторного района плюрипотентности Oct4, связанный 11 различными транскрипционными факторами [203].
Метилирование ДНК Метилирование - это ферментативная химическая модификация, добавление метильных групп (CH3) в специфических сайтах белков, ДНК и РНК. Одна из наиболее распространенных форм метилирования представляет собой превращение цитозина в 5метилцитозин в последовательности нуклеотидов CpG [204]. У человека и большинства млекопитающих ДНК-метилирование естественная модификация ДНК, и происходит только в CpG-динуклеотидах [205]. Метилирование может предотвращать расщепление ДНК в сайте узнавания фермента рестрикции. Реакция ДНКметилирования катализируется ферментом ДНК-метилтрансферазой, который осуществляет перенос метильной группы с S-аденозилметионина на цитозин, стоящий перед гуанином.
Метилирование - эпигенетический процесс, не меняющий последовательность ДНК [206, 207]. 70-80% всех CpG-динуклеотидов в геноме человека метилированы [205]. Некоторые гены, экспрессирующиеся в эмбриональном периоде, перестают функционировать к моменту рождения; профиль метилирования в тканях может меняться в течение жизни. Большинство CpG-островков (соответствующих промоторам) в норме не метилированы. Метилирование происходит, прежде всего, в районах генома с низкой плотностью CpG динуклеотидов.
Метилированные основания ДНК экспериментально обнаружены еще в 1948 году [208]. Метилированная ДНК высших эукариот содержит в основном 5-метилцитозин [204, 207, 209]. Существует несколько версий о роли метилирования ДНК: контроль экспрессии гена, контроль целостности хромосомы, контроль пре-рекомбинантных последовательностей (ретровирусных элементов) [210, 211].
Профиль метилирования, влияющий на функциональное состояние гена, передается в ряду клеточных поколений, в связи с этим развиваются методы определения возраста клеток в ткани - клеточного старения по данным метилирования [212].
Механизм инактивации гена посредством ДНК-метилирования функциями белков-метилаз [210]. Белок MeCp2 (methylated-DNA binding protein 2) связывается с метилированной ДНК и включается в комплекс, состоящий из гистоновых белков и деацетилазы [213]. Этот белковый комплекс, в свою очередь, инициирует компактизацию хроматина, что не дает связаться факторам транскрипции с промоторной областью и, следовательно, происходит инактивация гена [211].
Полагают, что метилирование промотора может быть одним из механизмов инактивации генов-супрессоров опухолевого роста в раковых клетках [149]. Список генов, инактивируемых через метилирование промоторной области, включает MyoD, Rb1, VHL, ген p16. Гиперметилирование промоторной области гена - рецептора эстрогенов (ER) обнаруживается в опухолях толстого кишечника [214]. Показано, что аберрантное метилирование промоторного района гена металлопротеиназы-3 (TIM-3) происходит в различных опухолях: раке молочной железы, раке толстой кишки, карциноме почки. В спорадических опухолях молочной железы показана инактивация посредством метилирования гена BRCA1, гена MYOD и гена ER [215, 216]. Таким образом, метилирование промоторной области может являться механизмом инактивации генов-супрессоров опухолевого роста.
В опухолях наиболее надежным методом для оценки частоты метилирования CpG-островков в настоящее время считается метод RLGS (restriction landmark genomic чувствительные рестриктазы (HpaII, HhaI, NotI, SacII, EagI, BssHII) с последующей амплификацией CpG-острова. Современный метод определения геномного метилирования основан на бисульфитной модификации ДНК с последующей метилспецифической амплификацией или секвенированием [212]. Метод основан на том, что бисульфит натрия преобразовывает все неметилированные цитозины в урацил, в то время как метилированные цитозины, стоящие перед гуанином остаются в не модифицированном состоянии.
1.3.2. Компьютерные методы распознавания регуляторных районов генов распознавания регуляторных районов генов эукариот. Компьютерный анализ геномных последовательностей дает возможность объяснить особенности структурнофункциональной организации известных районов геномов, позволяет предсказать функциональные сайты во вновь секвенированной геномной ДНК.
Стандарты описания функциональных сайтов При описании нуклеотидных последовательностей для обозначения классов нуклеотидов используется соответствующая номенклатура – 15-буквенный вырожденный код IUPAC (Таблица П3 в Приложении). Более точным способом представления и анализа выборок выровненных последовательностей длины L являются весовые матрицы размерности L 4. Элемент f(i,j) весовой матрицы F = |f(i, j)| определяет частоту встречаемости нуклеотида i (i =1,2,3,4 соответствует символам A, T, G и C) в позиции j (j = 1,.., L), подсчитанную по выборке выровненных нуклеотидных последовательностей. Оптимизированная весовая матрица W= |w(i,j)| может быть вычислена в логарифмической форме с учетом ожидаемых частот [166].
Участки последовательностей, сходство которых с весовой матрицей (мотивом) превышает пороговое значение, рассматриваются как потенциальные сайты связывания транскрипционных факторов [217]. Весовые матрицы ССТФ определены в базах данных, таких как TRRD [200], JASPAR [218], TRANSFAC [129], на основе компиляции результатов связывания ДНК с белковыми ТФ в экспериментах с помощью различных технологий.
Существуют другие способы представления оптимального расположения нуклеотидов в сайте для оценки силы связывания [219]. Традиционная весовая матрица (частотная матрица) может быть преобразована в позиционно-специфичная матрицу энергии связывания PSEM (Position Specific Energy Matrix) и обратно, используя экспоненциальную трансформацию [219, 220].
Методы компьютерного распознавания регуляторных районов Важнейшая задача анализа регуляторных районов – распознавание сайтов последовательности ДНК и по контекстным характеристикам нуклеотидной последовательности. Для обучения программ компьютерного распознавания и определения потенциальных сайтов связывания транскрипционных факторов разрабатываются базы данных регуляторных районов генов эукариот и ССТФ [129, 200]. Проблема распознавания сайтов связывания обусловлена тем, что хотя транскрипционные факторы связываются с ДНК специфично, большинство сайтов имеет лишь небольшую постоянную «коровую» (core) последовательность, составляющую 4- п.о., окруженную некоторым числом не постоянно встречающихся нуклеотидов [221].
Показано, что использование зависимостей между нуклеотидами, в частности динуклеотидных матриц позволяет значительно повысить эффективность распознавания сайтов [222].
Несмотря на то, что создано большое количество методов распознавания промоторов РНК-полимеразы II в геномах эукариот [221, 223-225], проблема повышения точности распознавания в целом остается нерешенной. Заметим, что большинство методов было разработано до начала массового секвенирования и обучалось на сравнительно небольших выборках данных.
При рассмотрении связывания ТФ с ДНК in vivo надо учитывать, что многие белковые факторы не работают по отдельности, часто формирую комплексы с другими факторами и таким образом, могут связывать ДНК прямо или опосредованно. В зависимости от архитектуры комплекса транскрипционных факторов, последовательности ДНК, связанные этим комплексом, могут казаться связанными в экспериментах ChIP-chip для каждого ТФ этого комплекса, хотя только один фактор связан с ДНК напрямую. Например, транскрипционные факторы дрожжей Mbp1 и Swi6, формируют MBF комплекс, играющий важную роль в регуляции клеточного цикла [226]. Swi6 связывает Mbp1, а Mbp1 контактирует с ДНК непосредственно, связываясь с последовательностью ACGCGT [227]. Таким образом, важным моментом анализа сайтов связывания является понимание возможности непрямого связывания ТФ с ДНК через другие белки, называемое также “piggy-back”.

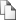
 [
[  «
«